Enhancer/Promoter Library 제작
Enhancer 및 promoter는 유전자 발현의 공간적 및 시간적 패턴을 조절하는데 중요한 역할을 하는 non-coding 게놈 영역입니다. 이러한 cis-regulatory element를 식별하고, 유전자 조절 활동을 분석하고, 새로운 조절 변이를 엔지니어링하는 것은 유전자 치료에 많은 영향을 미치는 유전자 발현을 이해하고 최적화하는데 중요합니다. VectorBuilder는 연구자 및 신약 개발자가 in vitro 및 in vivo에서 대규모 병렬 스크리닝을 통해 기능 functional enhancer/promoter를 효과적으로 식별할 수 있도록 종합적인 솔루션을 제공합니다.
디자인 서포트 의뢰하기 에서 무료 상담을 받아보세요.
중점 사항
- Streamlined full-service platform: Library 및 스크리닝 전략 디자인, library 제작, 바이러스 패키징, in vitro 및 in vivo 스크리닝, NGS 분석, hit identification 및 기능적 특성분석을 포함한 종합적인 솔루션.
- Expertise in library design: 강력하고, 편향되지 않고, 다양한 스크리닝을 위한 높은 signal-to-noise 비율, barcode 통합, NGS 판독 전략 등.
- High-complexity libraries with high-titer, high-purity virus packaging: Non-viral, lentiviral, AAV, piggyBac 및 기타 전달 시스템에서 사용 가능한 library (>108). 바이러스 패키징은 lentivirus (>108 TU/ml) 및 AAV (>1013 GC/ml)가 포함됩니다.
- Multiple in vivo models including non-human primate (NHP): 스크리닝에 사용할 수 있는 모델에는 mice, rats 및 두가지 NHP 종, crab-eating macaque (Macaca fascicularis, a.k.a. cynomolgus monkey) 및 rhesus macaque (Macaca mulatta)가 포함됩니다. 모든 동물 실험은 AAALAC-공인된 시설에서 수행됩니다.
서비스 워크플로우
다음은 enhancer/promoter 스크리닝의 일반적인 워크플로우입니다.
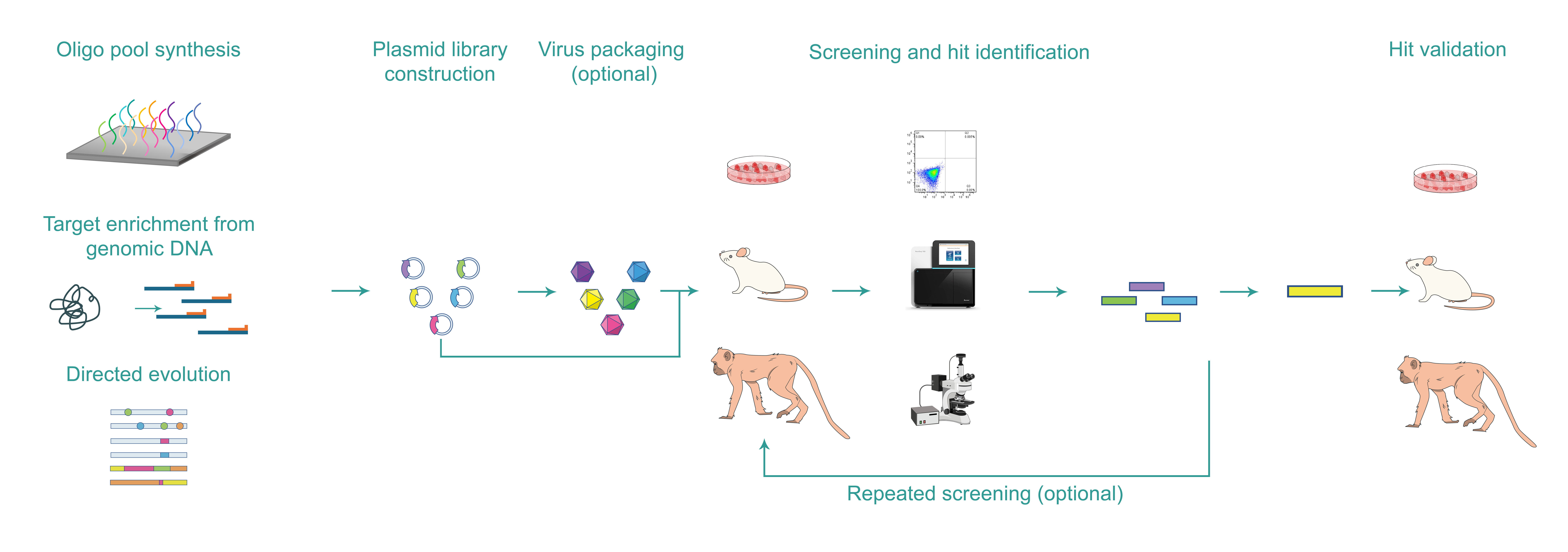
Enhancer/promoter library design and construction
In vitro/in vivo screening
Hit validation
Library 디자인
아래 그림은 enhancer (Figure 1) 및 promoter (Figure 2) library에 대한 일반적인 벡터 디자인을 보여줍니다:

Figure 1. Enhancer library를 위한 주요 벡터 구성요소.
Enhancer: 스크리닝할 enhancer 서열을 리포터 유전자의 상류 또는 하류에 삽입할 수 있습니다. Enhancer가 리포터 유전자의 하류(A)와 poly A signal 앞에 위치하면 리포터 유전자와 함께 전사되므로 mRNA-seq로 직접 측정할 수 있습니다. 리포터 유전자의 상류(B)에 위치할 때 바코드 서열은 일반적으로 리포터 유전자의 3' UTR 영역에 통합되고 리포터 유전자로 전사될 수 있습니다. 바코드는 enhancer의 대리 역할을 하며 mRNA-seq로 서열을 분석할 수 있습니다.
Minimal promoter: 사용자가 선택한 minimal promoter 서열이 여기에 배치됩니다. 전사를 활성화하기 위해 enhancer가 존재하는 경우 이는 다운스트림 리포터의 전사를 유도합니다. 이러한 enhancer가 없으면 minimal promoter는 거의 완벽하게 비활성화됩니다.
Click to view more information about commonly used minimal promoters
Kozak: Kozak consensus sequence. 이는 진핵생물에서 번역 개시를 용이하게 하는 것으로 생각되기 때문에 관심있는 ORF의 시작 코돈 앞에 배치됩니다.
Reporter: 시각적으로 검출 가능한 밝은 형광 단백질 유전자(예: GFP) 또는 화학발광 단백질 유전자(예: 루시퍼라제). 이를 통해 enhancer 활동을 매우 민감하게 감지할 수 있습니다.
Barcode: Enhancer 라이브러리 바코드는 개별 플라스미드 내의 고유하고 짧은 시퀀스입니다. 스크리닝 후 NGS 분석을 통해 각 바코드의 판독 횟수를 판별합니다. 고유한 바코드 서열을 특정 enhancer 변이체와 연관시킴으로써 특정 enhancer의 전사 활성을 확인할 수 있습니다.
SV40 late pA: Simian virus 40 late polyadenylation signal. 이는 업스트림 ORF의 전사 종료를 촉진합니다.
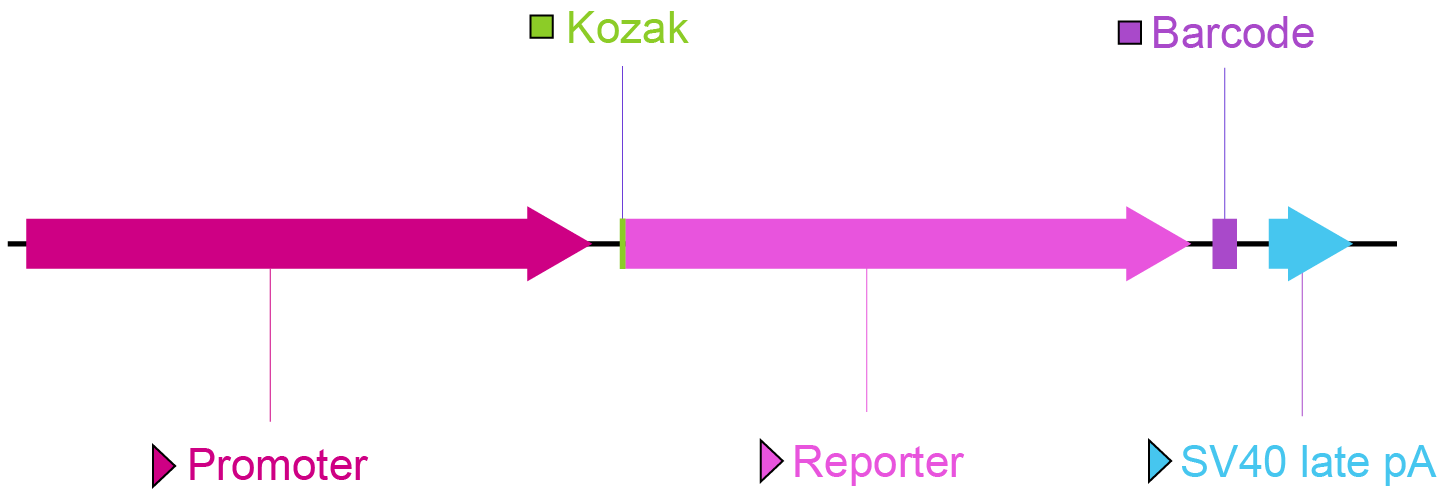
Figure 2. Promoter library를 위한 주요 벡터 구성요소.
Promoter: 스크리닝할 프로모터 서열을 여기에 삽입하여 리포터의 전사를 구동할 수 있습니다.
Kozak: Kozak consensus sequence. 이 구성요소는 진핵생물에서 번역 개시를 용이하게 하는 것으로 생각되기 때문에 관심 있는 ORF의 시작 코돈 앞에 배치됩니다.
Reporter: 시각적으로 검출 가능한 밝은 형광 단백질 유전자(예: GFP) 또는 화학발광 단백질 유전자(예: 루시퍼라제). 이를 통해 프로모터 활동을 매우 민감하게 감지할 수 있습니다.
Barcode: 프로모터 라이브러리 바코드는 개별 플라스미드 내의 고유하고 짧은 시퀀스입니다. 스크리닝 후 NGS 분석을 통해 각 바코드의 리드수를 판별합니다. 독특한 바코드 서열을 특정 프로모터 변이체와 연관시킴으로써 특정 프로모터의 전사 활성을 확인할 수 있습니다.
SV40 late pA: Simian virus 40 late polyadenylation signal. 이 구성요소는 업스트림 ORF의 전사 종료를 촉진합니다.
최적의 library 디자인은 성공적인 스크리닝의 핵심입니다. Enhancer/promoter library를 디자인할 때 고려해야할 몇 가지 중요한 사항은 다음과 같습니다:
- Delivery system
Non-viral plasmids, lentivirus 및 transposons은 in vitro 스크리닝에 널리 사용되는 반면, AAV는 in vivo 스크리닝에 자주 사용됩니다. 바이러스 titer와 serotype이 투여 경로 및 조직 친화성과 일치하는 것이 중요합니다. 또한 library의 복잡성이 높고 기능적 변이의 비율이 상대적으로 낮은 경우 non-viral plasmid transfection은 일반적으로 바이러스 또는 transposon 시스템보다 선호됩니다. 이는 세포 내 plasmid의 복제 수가 높아 효과적인 신호 판독을 촉진하기 때문입니다. 다양한 벡터 시스템 비교에 대한 자세한 내용은 FAQ를 참조하세요.
- Barcode
고유한 바코드의 통합은 주로 library 유형에 따라 다릅니다. Enhancer/promoter가 리포터 유전자의 상류에 배치되는 경우(Figure 1B 및 2), 바코드는 리포터 유전자의 하류인 3' UTR 앞에 위치해야 합니다. 스크리닝 후 mRNA가 추출되고 enhancer/promoter 활성의 대리 역할을 하는 바코드가 NGS를 사용하여 판독되어 해당 enhancer/promoter의 전사 활성을 평가할 수 있습니다. 반대로 enhancer가 리포터 유전자의 하류에 위치하면(Figure 1A) NGS를 통해 그 활성을 직접 측정할 수 있습니다. 그러나 enhancer가 긴 경우 정확한 판독을 보장하기 위해 바코드를 통합해야 할 수도 있습니다. 바코드에 대한 자세한 내용은 Barcode Library 제작 페이지를 참조하세요.
- Scalability
겟 세포에 대한 cell sorting을 수행하면 기능적 enhancer/promoter 검출의 효율성이 향상되어 스크리닝 프로세스의 확장성과 효율성이 향상되는 경우가 많습니다. Cell sorting은 알려진 세포 마커를 사용하거나 library 컨스트럭트에서 리포터 유전자의 발현을 검출함으로써 달성될 수 있습니다.
Library 제작
Enhancer/promoter 단편은 보통 아래에 언급된 일반적인 접근법을 사용하여 얻을 수 있습니다:
- Oligo pool synthesis
Chip-based oligo 합성은 미리 설계된 짧은 enhancer/promoter variants를 생성하는데 이상적입니다. 이 방법은 낮은 오류율로 최대 300 nucleotides 길이의 올리고 풀을 합성할 수 있습니다.
- Target enrichment from genomic DNA
타겟 게놈 영역의 단편은 probe hybridization을 통해 사이즈 선택, 절단된 게놈 DNA 또는 BAC (bacterial artificial chromosome)에서 캡처될 수 있습니다. 또는 편향 없는 shotgun 방법을 활용하여 전체 게놈 또는 BAC DNA를 포함할 수 있습니다.
- Directed evolution
Error-prone PCR, degenerate codon 통합 또는 DNA shuffling을 포함한 다양한 mutagenesis 전략을 사용하여 알려진 enhancer/promoter로부터 새로운 서열 변이체를 개발할 수 있습니다.
In vitro/in vivo 스크리닝
Enhancer/promoter variants의 스크리닝은 in vitro 및 in vivo에서 수행될 수 있습니다. In vitro 선택은 일반적으로 신속하고 기술적으로 간단하지만, in vivo에서 발견되는 생리학적 조건을 복제할 수는 없습니다. In vivo 동물 모델은 향상된 전사 활성을 갖는 enhancer 및 promoter를 스크리닝하기 위한 신뢰할 수 있는 플랫폼을 제공합니다. 마우스와 NHP (non-human primates) 모두 enhancer/promoter library의 in vivo 선택에 활용되지만, NHP 모델은 유전자 치료제 응용 분야에 중요한 인간과의 높은 유사성으로 인해 원하는 특성을 가진 조절 요소를 식별하기 위한 보다 임상적으로 관련 있는 플랫폼을 제공합니다.
in vivo 스크리닝의 경우 tail vein injection, facial vein injection (neonatal mice 및 rats의 경우), intracerebroventricular injection, intrathecal injection, subretinal injection, intravitreal injection, intratympanic injection, intramuscular injection 등을 포함한 다양한 투여 경로를 사용할 수 있습니다.
Enhancer/promoter library를 타겟 세포 또는 동물에 전달한 후, 리포터 유전자 또는 바코드의 발현을 측정하여 후보 조절 요소 변이체의 활성을 평가할 수 있습니다. 원하는 특성을 가진 조절 요소를 확인하기 위해 활성이 향상된 변이체는 여러 차례의 스크리닝을 추가로 진행할 수 있습니다. Figure 3은 cone-specific promoter AAV library를 구성하고 in vivo 스크리닝을 위한 주요 단계를 보여줍니다.
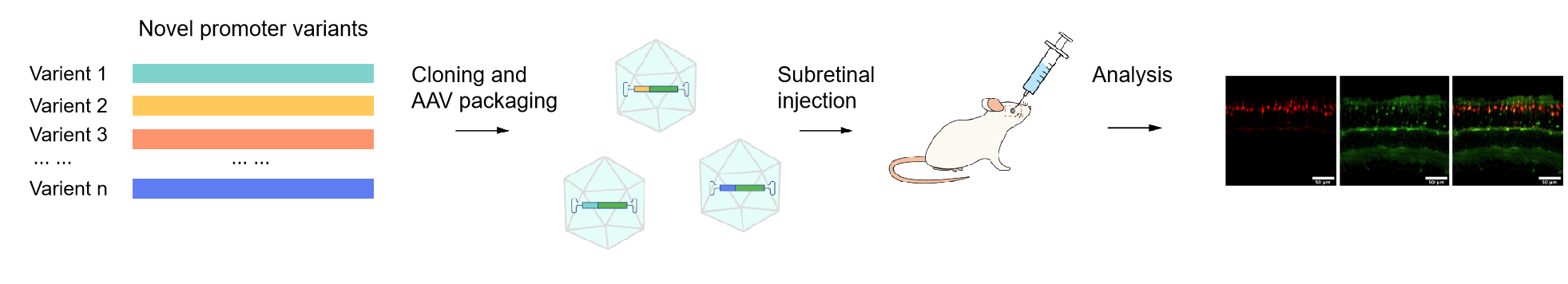
Figure 3. Cone-specific promoter AAV library 제작 및 스크리닝. (A) 실험 워크플로우. 새로운 변종은 알려진 광수용체 특이적 프로모터로부터 DNA 셔플링을 통해 생성되었습니다. 그런 다음 새로운 변종을 AAV 백본에 클로닝하여 GFP 리포터의 발현을 유도했습니다. AAV 프로모터 라이브러리를 마우스의 망막하 영역에 주입하였다. Cone-specific promoter에 대한 스크리닝에는 형광 이미징, 세포 분류 및 NGS 시퀀싱이 포함되었습니다. (B)라이브러리의 망막하 발현 패턴. GFP를 구동하는 AAV 프로모터 라이브러리는 TdTomato를 구동하는 ProA1 프로모터를 운반하는 다른 AAV와 함께 주입되었습니다. ProA1은 알려진 Cone-specific promoter입니다. 빨간색과 녹색 형광의 공동 위치화는 프로모터 라이브러리에 Cone-specific promoter가 존재함을 나타냅니다.
Hit validation
Enhancer/promoter 스크리닝에서 확인된 positive hits는 in vitro 또는 in vivo에서 원하는 활성에 대한 추가 특성 분석이 필요합니다. 특성 분석을 위해 기능적으로 가장 관련성이 높은 시스템을 사용하는 것이 좋습니다. 예를 들어, Figure 3에 설명된 cone-specific promoter 스크리닝의 경우 확인된 새로운 promoter variants는 마우스와 NHP의 특이성과 강도를 결정하기 위해 특성화되었습니다. 아래 Figure 4는 새로운 VectorBuilder(VB) photoreceptor cone cell-specific promoter의 기능적 검증을 보여줍니다.
Figure 4. 스크리닝을 통해 확인된 새로운 VB promoter의 기능적 특성 분석. (A) 마우스에서 VB promoter에 의해 구동되는 nucleus-localized mCherry의 cone-specific 발현. mCherry를 구동하는 VB promoter를 운반하는 AAV8을 subretinal injection 했습니다. Cone cell은 cone-cell marker인 Arrestin-C(green)로 표지 되었습니다. (B) Crab-eating monkey에서 VB promoter에 의해 구동되는 mCherry의 cone-specific 발현. mCherry를 구동하는 VB promoter를 운반하는 AAV8을 subretinal injection으로 투여했습니다.
주문 방법
고객이 제공하는 library plasmid pool
고객이 제공하는 플라스미드 풀을 사용하는 경우 시료 제출 가이드라인에 따라 준비하여 보내주십시오. 시료의 손상이나 배송 지연을 방지하려면 배송 가이드라인을 따라 주시기 바랍니다. 고객이 제공하는 시료는 당사의 필수 QC를 거치며 각 항목에 대해 추가 $100의 요금이 부가될 수 있습니다. 고객이 제공한 시료는 QC를 통과한 후 생산이 시작됩니다. 고객이 제공한 플라스미드 풀에 대해서는 라이브러리의 복잡성이나 균일성에 대해 보장하지 않습니다.


