코돈 최적화
코돈 최적화는 유전자를 heterologous (다른 숙주에서) 발현할 때, 유전자를 클로닝할 때 문제가 발생하는 경우, 또는 유전자 발현 레벨을 최적화할 때 유용한 도구입니다. VectorBuilder의 코돈 최적화 도구는 Design Studio 에 내장되어 있으며, 여기에서 독립적으로도 사용할 수 있습니다.
대부분의 아미노산은 여러 코돈에서 번역될 수 있지만, 코돈 편향은 하나의 코돈이 다른 코돈보다 선호도를 반영하며 이는 종 마다 다릅니다. 이것은 한 유전자가 다른 숙주 종에서 번역이 감소될 수 있습니다. 이 도구는 숙주 유기체의 코돈 편향을 활용하여 더 높은 효율로 동일한 아미노산 서열을 생성하여 codon adaptation index (CAI)를 최적화할 수 있습니다.
코돈 최적화는 관심 유전자의 클로닝 효율을 향상시키기 위해 사용될 수 있습니다. 이 도구는 GC 함량 및 반복 서열을 최적화하고, mRNA 안정성을 개선하고, 제한효소 인식 부위를 회피함으로써 전사 또는 번역 효율을 개선하는데 도움이 될 수 있습니다.
아래에 VectorBuilder 코돈 최적화 도구의 다양한 기능들에 대한 예들을 보여드립니다.
1. Optimizing sequences for codon usage in desired target species View more
Figure 1 은 코돈 최적화 도구를 이용하여 Trichoplusia ni 에서 유래된 piggyBac transposase의 염기 서열을 인간의 코돈으로 최적화했을 때 CAI가 0.93이 되는 것을 보여 주고 있습니다. 코돈 최적화를 하기 전에는 CAI가 0.69였습니다. 해당 종의 CAI는 그 종에서 높은 수준으로 발현되는 유전자들에서 선호되는 코돈의 사용 빈도를 정량화한 것으로, 0 과 1 사이의 값을 갖습니다. 특정한 종을 위한 유전자의 CAI가 높을수록 그 종에서 최적으로 발현될 가능성이 높아집니다.
Figure 1. VectorBuilder의 코돈 최적화 도구를 사용한 타겟 종에 맞춘 코돈을 위한 염기 서열 최적화.
2. Optimizing sequences with high GC content View more
Figure 2 GC 함량이 높은 염기 서열의 최적화 – Figure 2는 코돈 최적화 도구를 사용하였을 때69.3% 였던 생쥐의 Hoxa4 유전자의 전체 GC 함량이 59.5%로 감소한 것을 보여 주고 있습니다. 클로닝 과정에서 유전자를 합성해야 하는 경우에 유전자 합성 성공 가능성을 높이기 위해서는 GC 함량이 60% 정도가 가장 좋습니다.
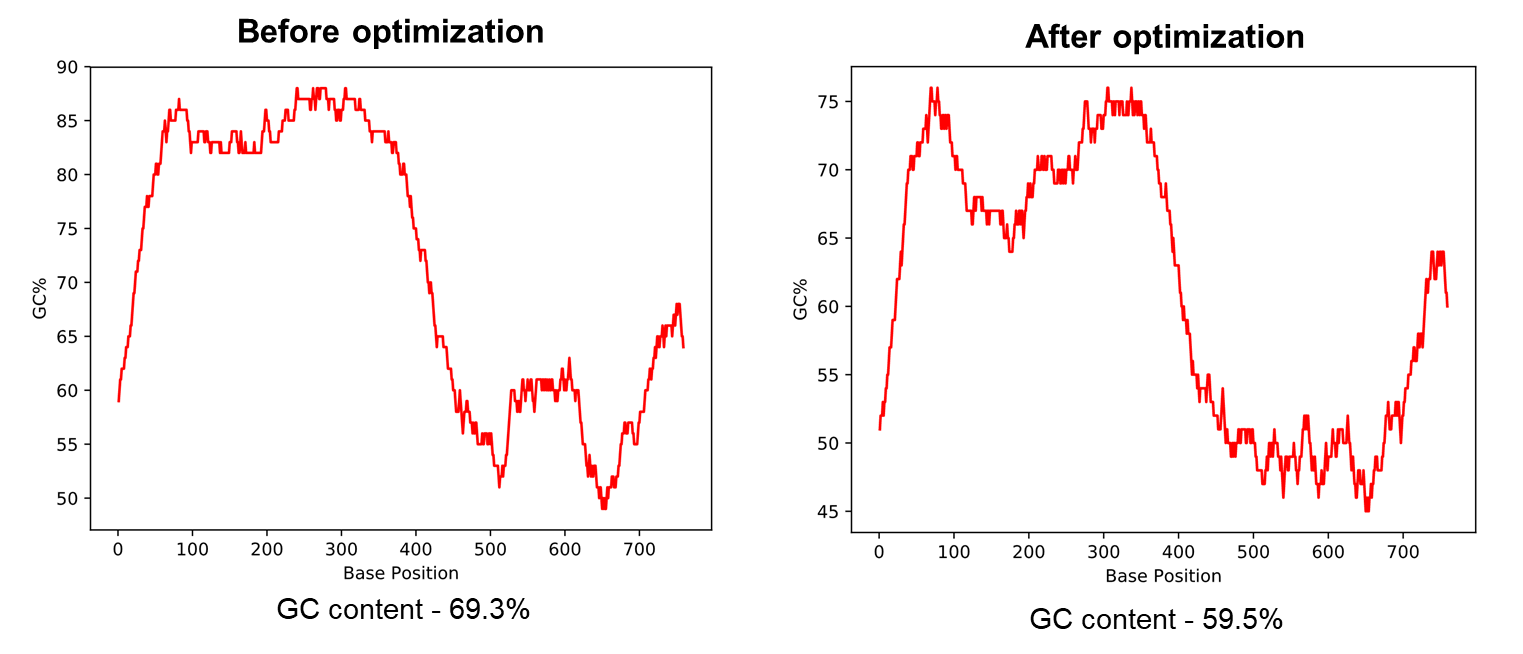
Figure 2. VectorBuilder의 코돈 최적화 도구를 사용한 높은 GC함량을 가진 염기 서열의 최적화.
3. Optimizing sequences with repetitive regions View more
Figure 3 은 코돈 최적화 도구를 사용한 최적화 전후의 인간 immunoglobulin heavy chain 유전자의 염기 서열을 비교한 dot plot들입니다. 최적화하기 전의 dot plot에서는 여러 개의 대각선들로 표시되는 것처럼 반복 서열이 많이 존재하는 것을 볼 수 있지만, 최적화를 한 후에는 이러한 반복되는 서열들이 많이 줄어든 것을 dot plot에서 볼 수 있습니다.
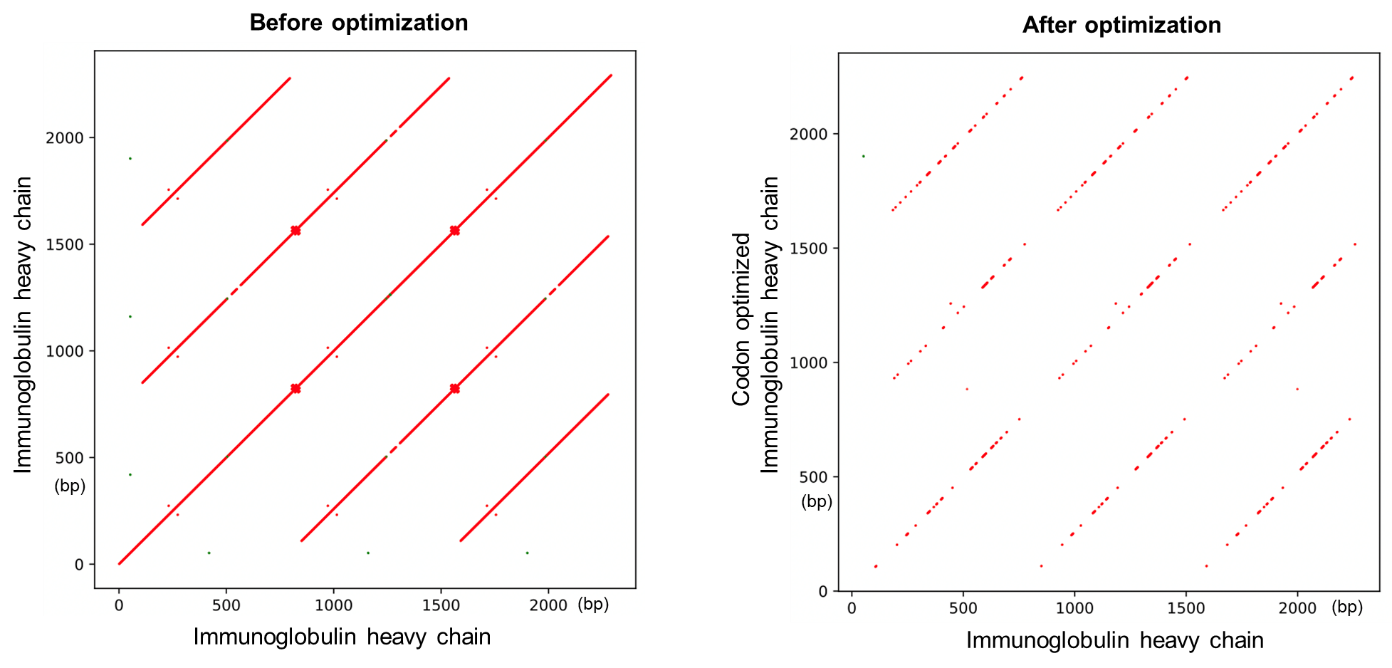
Figure 3. VectorBuilder의 코돈 최적화 도구를 사용한 반복되는 염기 서열들의 감소.
Protein production
In order to produce proteins, a cell must first translate the relevant mRNA strand. Following transcription, the mRNA exits the nucleus where each group of three nucleotides is matched to a tRNA molecule carrying an amino acid (Figure 1A). These groups of 3 nucleotides are codons, and each corresponds to an amino acid. Because there are only 20 amino acids and many more possible combinations of nucleotides, there is redundancy in this code (Figure 1B).


Figure 1. Formation of a protein through transcription and translation (A) of codons. Each codon corresponds to an amino acid or direction (start/stop).
Codon bias
Although there are multiple options for making each amino acid, their usage is not based on chance. This is because each species exhibits codon bias, the preference for making an amino acid with a certain codon. For instance, alanine (Ala) is coded by GCU, GCC, GCA, and GCG (Figure 1B), but in humans, GCC is used about 40% of the time. Different organisms have different codon preferences, which influences RNA processing and therefore protein folding and function. This creates complications when expressing one gene in another organism, i.e. heterologous gene expression.
The Codon Adaptation Index (CAI) is a measure of how well given codons match with the biases of an organism, ranging from 0 to 1. A CAI of 1 reflects a coding sequence where all amino acids reflect the most frequently used codons in that organism. Our Codon Optimization tool presents a sequence that balances an optimal CAI with other factors that can influence molecular experiments.
Enhancing cloning efficiency
Codon optimization can also aid in increasing cloning efficiency based on the distribution of nucleotides across the sequence. GC content is an important variable to consider when designing and troubleshooting experiments. If GC content is too high or too low, stability of the query sequence is negatively affected. Our GC Content Calculator tool allows for independent GC analysis over an entire sequence and within segments of a sequence. However, our Codon Optimization tool incorporates this analysis to optimize this variable by finding synonymous codons that increase or decrease GC content as needed.
Additionally, sequences that have a high frequency of repeats can present complications in cloning efforts due to the lack of unique primer binding sites, and sequences with recognition sites for restriction enzymes can present challenges in experimental design. Using our Codon Optimization tool allows for all of these factors to be optimized in unison with codon bias to provide a sequence that is most likely to efficiently produce your protein in your system.
- Sequences in both GenBank and FASTA formats can be recognized.
- You can input a DNA/RNA sequence or protein sequence.
- DNA/RNA sequences must begin with start codon ATG and must be in a multiple of 3 for a complete codon sequence.


