이중 gRNA (Dual-gRNA) CRISPR Library
VectorBuilder는 인간 및 마우스 세포에서 CRISPR 기반의 전체 유전체의 knockout screening을 위한 이중 gRNA 렌티바이러스 library를 제공합니다. 가능하면 각각의 유전자는 별개의 벡터로부터 4~6개의 서로 다른 gRNA 쌍에 의해 중복으로 타겟팅되도록 하였습니다. 이러한 library는 genome-wide loss-of-function screening을 수행하기 위한 강력하면서 비용면에서 효율적인 도구입니다. 또한 고객의 타겟 유전자 리스트에 대한 맞춤형 pooled gRNA library를 설계하고 제작할 수 있습니다.
제공되는 이중 gRNA CRISPR library의 유형
- 전체 인간 유전체 (20,048 인간 유전자) 를 타겟팅하는 ready-to-use, pooled, 렌티바이러스 library
- 전체 마우스 유전체 (20,493 마우스 유전자) 를 타겟팅하는 ready-to-use, pooled, 렌티바이러스 library
- 맞춤형 pooled gRNA library (E.coli stock, DNA 또는 재조합 바이러스)
추천글 보기
주문에 관련된 정보 Price Match
| Product Name | No. of Genes | No. of gRNA Pairs | Scale | Catalog No. | Price (USD) | Turnaround | Buy Now |
|---|---|---|---|---|---|---|---|
| Human Whole-Genome Dual-gRNA Library | 20,048 | 91,926 |
Medium (>1.0x108 TU/ml, 1 ml) |
LVM(Lib190505-1046fgb) | $2,999 | 7-14 days | |
|
Plus (>1.0x108 TU/ml, 5 ml) |
LV5M(Lib190505-1046fgb) | $3,499 | |||||
| Mouse Whole-Genome Dual-gRNA Library | 20,493 | 90,344 |
Medium (>1.0x108 TU/ml, 1 ml) |
LVM(Lib190505-1050kpm) | $2,999 | ||
|
Plus (>1.0x108 TU/ml, 5 ml) |
LV5M(Lib190505-1050kpm) | $3,499 |
당사의 premade 이중 gRNA CRISPR library가 고객의 수요를 만족시키지 못하는 경우, 당사의 custom library 제작 서비스를 보시기 바랍니다.
Deconvolution 서비스
CRISPR library screening을 수행한 후 NGS에 의한 gRNA hits을 확인하는 방법에 대한 확신이 없으신가요? 고객의 genomic DNA를 VectorBuilder로 보내주시면 NGS library를 만들고 high-throughput sequencing을 수행한 후, 데이터를 분석하여 수 주 이내에 각 샘플의 정확한 gRNA 카운트를 제공해드립니다.
Deconvolution 서비스를 원하시는 경우 디자인 의뢰하기로 문의사항을 보내주시기 바랍니다.
기술적인 정보
제품의 특징 View more
고유한 이중 gRNA 렌티바이러스 벡터 디자인
상용화된 단 하나의 전체 유전체에 대한 이중 gRNA 렌티바이러스 library로, library의 각 렌티바이러스 벡터에는 한 쌍의 gRNA 발현 카세트와 EGFP/puromycin 이중 marker 카세트가 포함되어 있습니다 (Figure 1). 동일한 벡터에서 쌍을 이룬 gRNA들은 벡터가 세포에 도입될 때 동시에 발현되면서 동일한 유전자에 있는 두개의 개별 부위를 타겟팅함으로써, 두개의 CRISPR 절단 부위를 생성하여 knockout screening을 위한 넓은 범위의 loss-of-function deletion이 일어나도록 합니다. 각 벡터에 있는 두개의 gRNA들은 두개의 서로 다른 U6 promoter (인간 U6 및 macaque U6) 및 두개의 서로 다른 gRNA 스캐폴드 (Nat Methods. 14:573 (2017))의 결합으로 생성됩니다. 이는 두 gRNA cassette 사이의 원치 않는 재조합을 감소시키고, library로 transduction된 세포에서 upstream, downstream gRNA의 PCR 증폭 및 NGS가 가능하도록 합니다.
Figure 1. 이중 gRNA library 벡터의 map.
이중 gRNA library 벡터의 상세한 map과 서열을 원하시면 여기를 눌러주시기 바랍니다.
전체 유전체에 대한 high-coverage 타겟팅
인간 및 마우스 이중 gRNA 렌티바이러스 library는 각각 20,048개의 인간 유전자와 20,493개의 마우스 유전자를 타겟으로 합니다. 평균적으로 각각의 유전자는 다음과 같은 조건들에 의하여 선별된 4-6개의 상이한 gRNA 쌍에 의해 타겟팅됩니다: 1) off-target 효과를 최소화하기 위한 off-target 스코어, 2) 두 부위에 걸친 deletion이 유전자 기능의 상실로 이어질 가능성이 높게 만들기 위한 절단 부위의 위치, 3) 각 절단 부위의 국소 변이보다는 절단 부위 사이의 큰 deletion이 일어나도록 하는 절단 부위 사이의 거리, 4) knockout되는 전사체의 수를 최대화하기 위한 각 유전자에서 knockout의 영향을 받는 alternative transcript의 수. 모든 gRNA 절단 부위는 exon으로 확인된 서열 내에 위치합니다. 따라서 절단 부위 사이의 큰 deletion이 생성되지 않더라도 절단 부위 내의 국소 변이 (일반적으로 작은 deletion) 로도 유전자의 기능을 잃게 할 가능성이 있는 변이를 생성할 수 있습니다. 타겟 유전자 및 gRNA의 상세한 리스트는 “문서 자료”에서 확인하실 수 있습니다.
NGS에 의한 library 품질의 검증
이중 gRNA 렌티바이러스 CRISPR library는 NGS에 의해 쌍을 이룬 두 gRNA들의 서열 분석을 완전히 함으로써 검증되었습니다. 이에 의하여 전체 sequencing reads의 >85% 및 >75%가 각각 인간 및 마우스 library에 대해 디자인된 gRNA 들과 일치함을 보여주었습니다. 또한 인간 및 마우스 library에 대해 디자인된 gRNA 쌍의 >99% 및 >97%가 각각 NGS에 의해 검출되었습니다. 따라서 두 library 모두 디자인된 gRNA 쌍들을 낮은 오류율로 매우 잘 포함하고 있음을 확인할 수 있었습니다. 이는 현재까지 보고된 최고 품질의 이중 gRNA library로 생각됩니다.
높은 균일성
두 library 모두 gRNA 쌍의 representation이 매우 균일함을 보여주고 있습니다 (Figure 2).
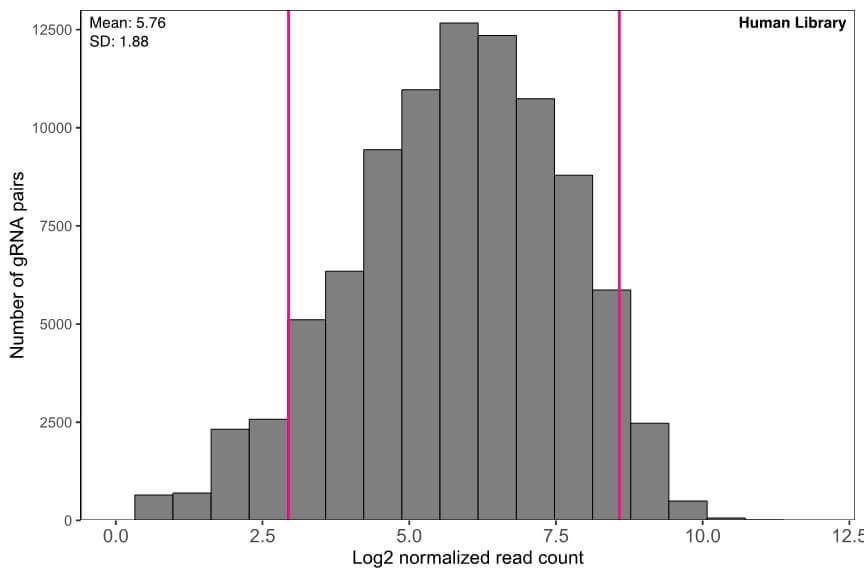
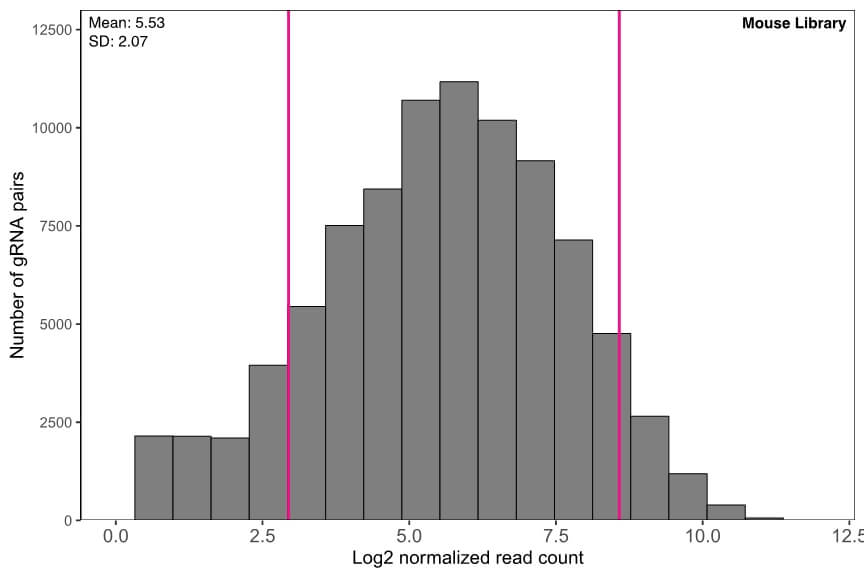
Figure 2. 서로 다른 pooled library의 gRNA pair representation. gRNA read count를 NGS library size (10 million reads)로 normalization한 후 log2 scale로 나타내었습니다.
잘 정립된 렌티바이러스 시스템과 바로 사용할 수 있는 높은 titer의 렌티바이러스
Pooled CRISPR library는 3세대 렌티바이러스 벡터 시스템에서 발현되며, 이는 다양한 세포에서 타겟 유전자의 발현을 안정적으로 knockdown할 수 있는 고효율 시스템입니다. 이 렌티바이러스 벡터 시스템은 gRNA를 세포에 영구적이고 효율적이며 균일하게 도입할 수 있기 때문에 in vitro 유전자 screening에 이상적입니다. 모든 pooled CRISPR library는 높은 functional titer (>10^8 TU/ml)의 바로 사용 가능한 렌티바이러스로 제공되어 고객이 바이러스 패키징 및 titer를 측정하는데 소요되는 시간을 절약해드립니다. 3세대 렌티바이러스 벡터 시스템은 자가복제 능력이 무력화되어 향상된 생물학적 안전성을 가지도록 최적화되었습니다.
효율적이며 다양한 선별 또는 transduction된 세포의 추적을 위한 이중 marker
EGFP 및 puromycin 내성 유전자 (Puro)의 이중 marker 발현 카세트가 렌티바이러스 벡터에서 발현되어 puromycin에 의하여 transduction된 세포를 선별하고 녹색 형광에 의하여 시각적인 추적을 가능하게 합니다 (Figure 3).

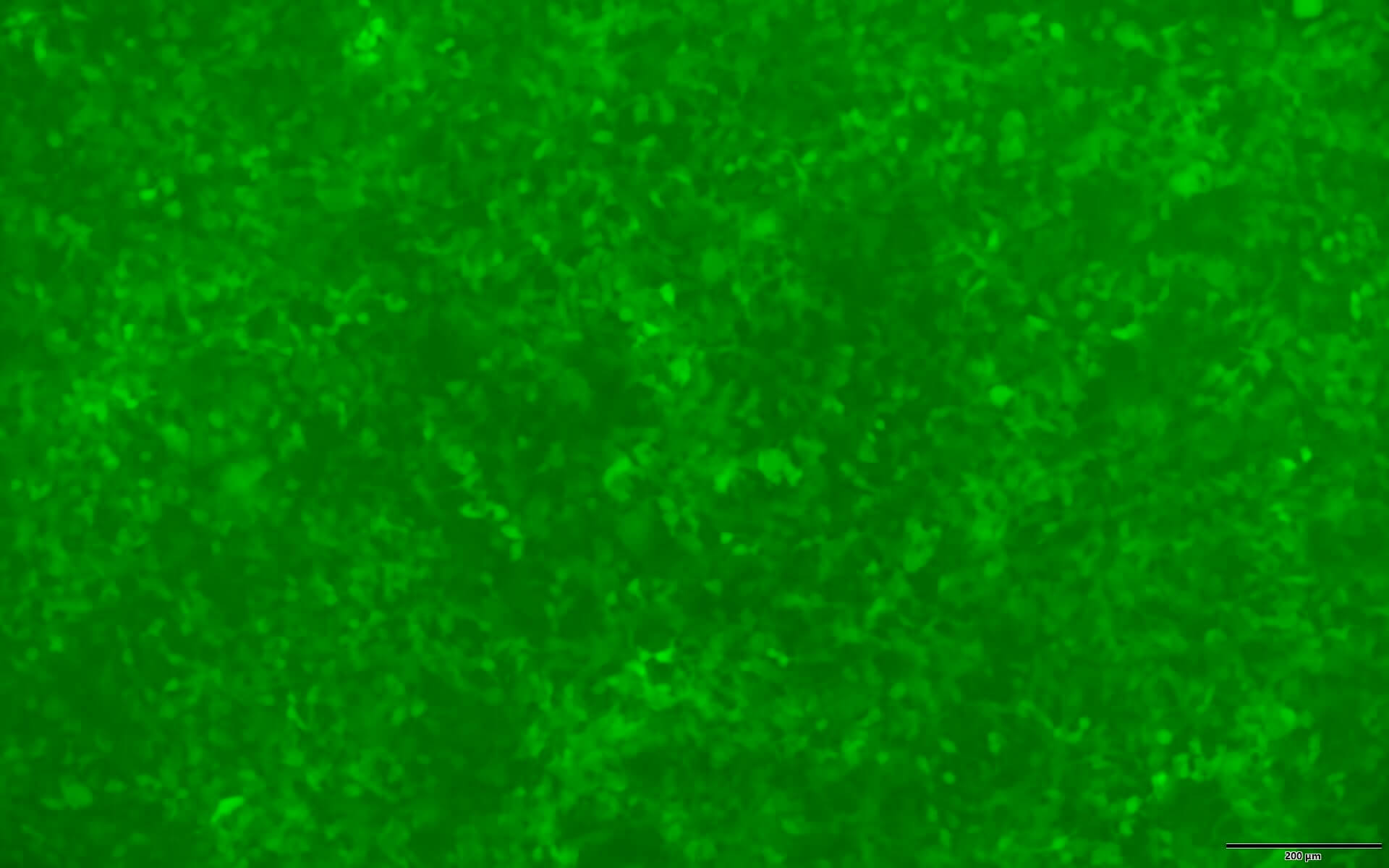
Figure 3. 인간 Whole Genome 이중 gRNA 렌티바이러스 Library (MOI=10)로 transduction한 293T 세포들을 puromycin (1.5 ug/ml)으로 4일간 선별한 후의 EGFP 발현. 왼쪽: bright field. 오른쪽: EGFP.
Pooled CRISPR library를 사용하는 genetic screening의 워크 플로우 View more
Pooled 이중 gRNA 렌티바이러스 library를 이용하는 CRISPR/Cas9 screening의 일반적인 워크플로우는 아래 Figure 4에서와 같습니다. 먼저 Cas9을 발현하는 세포들을 렌티바이러스 library로 transduction하고 transduction된 세포를 렌티바이러스 벡터에 존재하는 marker 유전자 (항생제 내성 또는 형광 marker) 의 발현에 의하여 선별합니다. 그 다음에, transduction된 세포를 기준 집단과 실험 집단으로 분할하고, 실험 집단에 특정한 selective pressure (약물 처리 또는 반복 계대 배양 등)를 적용하여 관심있는 표현형을 나타내는 세포를 확인합니다. Screening 전략에는 세가지 주요 유형이 있습니다. 1) selective pressure에 노출되었을 때 생존하는 세포에서 강화되거나 (enriched) 고갈되는 (depleted) gRNA를 검색하는 viability screening; 2) 세포 내에서 높거나 낮은 reporter의 발현 수준에 따라 강화되는 gRNA를 검색하는 reporter screening (예를 들어 reporter 유전자 발현을 조절하는 전사 인자를 타겟팅하는 gRNA); 3) cell invasion, migration 등과 관련된 유전자에 영향을 미치는 gRNA를 확인하는 behavior screening. Screening 후 실험 및 기준 세포들로부터 Sanger sequencing 또는 NGS에 의하여, 기준 그룹과 비교하여 실험 그룹에서 강화되거나 고갈된 gRNA를 확인합니다. 강화되거나 고갈된 gRNA에 연관된 잠재적인 타겟 후보 유전자들은 downstream functional study에 의한 추가 연구로 확인할 수 있습니다.
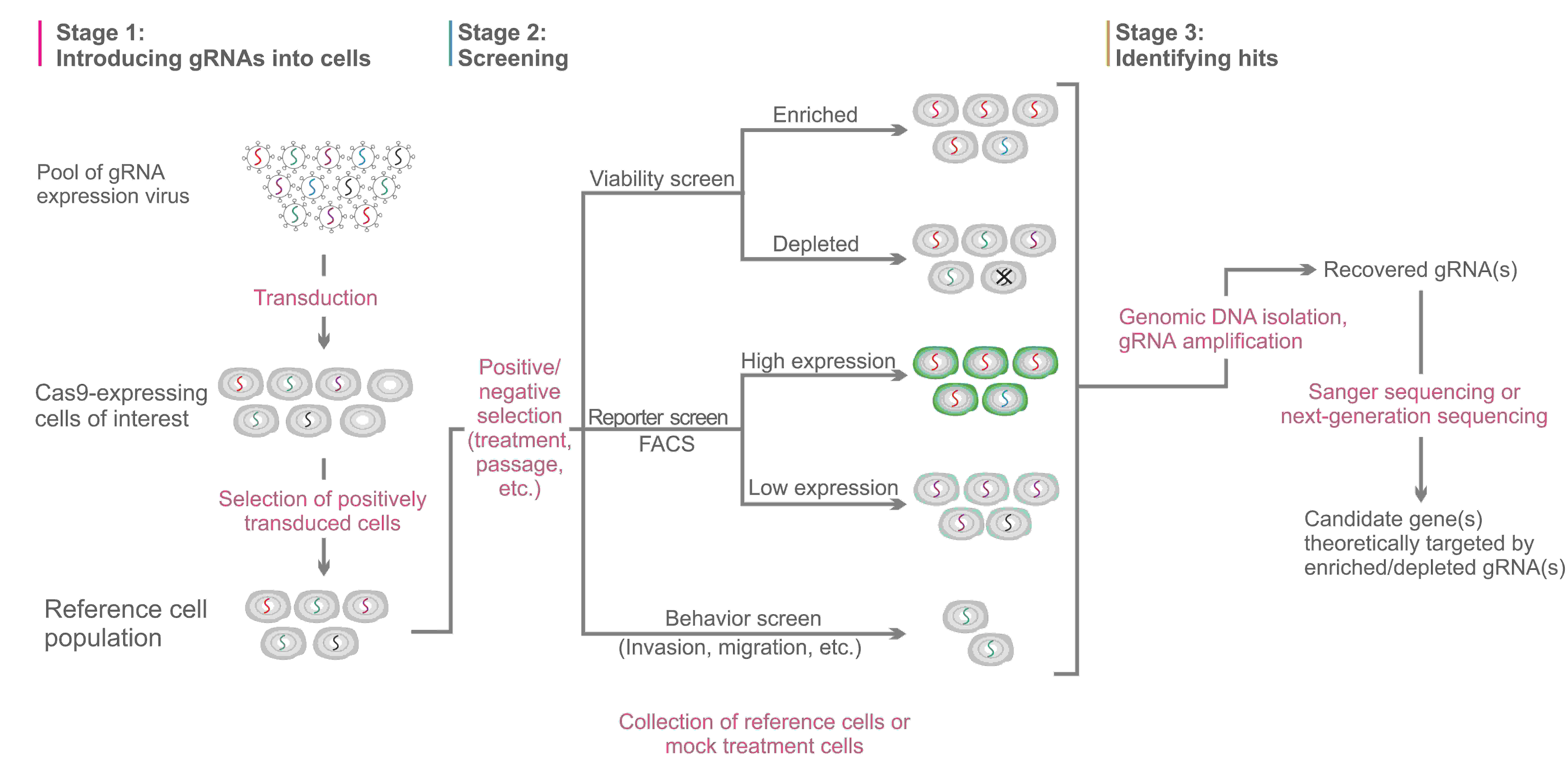
Figure 4. Pooled 이중 gRNA 렌티바이러스 library를 이용한 CRISPR 기반의 knockout screening의 워크플로우. [Acta Biochim Biophys Sin 44:103-112 (2012)에서 인용함]
문서 자료들 View more
타겟 유전자 및 shRNA의 전체 리스트
자주 묻는 질문
단일 gRNA library와 비교할 때 이중 gRNA library의 장점은 무엇입니까?
이중 gRNA library는 넓은 범위의 deletion이 일어나도록 함으로써 loss-of-function을 위한 변이를 훨씬 효율적으로 도입하기 때문에 knockout screening을 위해서는 단일 gRNA library에 비하여 훨씬 강력합니다. 이중 gRNA library의 각각의 CRISPR 벡터는 같은 유전자를 타겟팅하는 한 쌍의 gRNA를 포함하고 있습니다. Cas9을 발현하는 세포에 도입되었을 때, 각각의 벡터는 같은 타겟 유전자에 두개의 절단 부위를 만듭니다. 세포가 절단 부위 복구를 시도하는 중에 두 절단 부위를 포함하는 대규모의 deletion이 일어나게 됩니다. 두 절단 부위는 기능적으로 중요한 영역을 포함하도록 디자인되어 있기 때문에 이 부분을 포함하는 deletion이 일어나게 되면 대부분 유전자의 기능을 잃게 됩니다.
CRISPR knockout screening을 위해서 1-벡터 시스템과 2-벡터 시스템 중 어느 쪽을 사용하는 것이 더 좋습니까?
일반적인 knockout screening을 위하여, pooled CRISPR library는 1-벡터 또는 2-벡터 시스템으로 구성이 될 수 있습니다. 1-벡터 시스템의 경우 Cas9이나 변형 Cas9이 gRNA와 같은 벡터에서 발현이 되고, 2-벡터 시스템의 경우 Cas9이나 변형 Cas9이 gRNA와 별개의 벡터에서 발현됩니다. 또한 2-벡터 시스템에서는 gRNA를 발현하는 벡터가 Cas9을 안정하게 발현하는 세포에 도입될 수도 있습니다. 1-벡터 시스템의 장점은 두가지 별개의 벡터를 함께 transfection/transduction할 필요가 없고, Cas9을 안정적으로 발현하는 세포주의 존재에 따른 제한을 받지 않습니다. 하지만, 2-벡터 시스템에 비하여 다양성이 좀 떨어지고 클로닝과 바이러스 패키징이 덜 효율적이라는 단점도 있습니다.
gRNA specificity score는 어떻게 계산됩니까?
VectorBuilder는 CRISPR library design(CLD)에 사용된 알고리즘을 따라 gRNA의 특이성 스코어를 계산합니다. 간략하게, 하나의 생물종에 대한 주어진 gRNA가 N(20)NGG 서열을 타켓팅한다고 할 때, 그 생물종의 유전체 서열 내에서 타켓 서열과 3개 이하의 mismatch를 갖는 모든 잠재적인 off-target 서열들을 검색하고, 각각의 잠재적인 off-target 부위에 대하여 off-target score를 계산합니다. 모든 off-target 부위의 score를 종합하여 최종적인specificity score를 계산하며, 이 score는 0과 100 사이의 값으로 score가 더 클수록 더 높은 targeting specificity를 의미합니다.
Specificity score는 대략적인 가이드에 불과하다는 점에 유의하시기 바랍니다. 실제 타켓팅 효율과 정확성은 score에 의해 예상할 수 있는 것과 차이가 있을 수 있습니다. 또한 score가 낮은 gRNA들도 여전히 잘 작용할 가능성이 있습니다.


