mRNA 유전자 전달 솔루션 (mRNA Gene Delivery Solutions)
VectorBuilder는 백신, 유전자 편집, chimeric antigen receptor (CAR), 세포 또는 배아에서의 단백질 발현과 같은 mRNA 기반 치료제 개발을 위한 원스톱 솔루션을 제공합니다. 광범위한 디자인 및 생산 경험을 바탕으로 mRNA 기반 백신 및 유전자 치료 개발을 가속화 할 수 있도록, in vitro transcription (IVT) 벡터 디자인, 벡터 클로닝, in vitro mRNA 합성, mRNA-LNP 생산 및 in vitro/in vivo 기능 테스트를 지원하고 있습니다. mRNA 치료제의 대량 생산에 대해서는 CDMO 서비스를 참조하시기 바랍니다. 바로 사용 가능한 mRNA 제품은 IVT mRNA 및 LNP-mRNA 를 확인하시기 바랍니다.
서비스 세부 사항
 IVT vector design & cloning
IVT vector design & cloning
- 상업적 사용을 위한 IP 제약이 없는 royalty-free IVT 백본
- 높은 발현을 달성하기 위한 서열 최적화
- 5’ and 3’ UTR
- 코딩 서열
- Kozak
- 높은 발현을 위한 120nt(및 더 긴) polyA tail의 강력한 클로닝 및 전사
 IVT mRNA & LNP manufacturing
IVT mRNA & LNP manufacturing
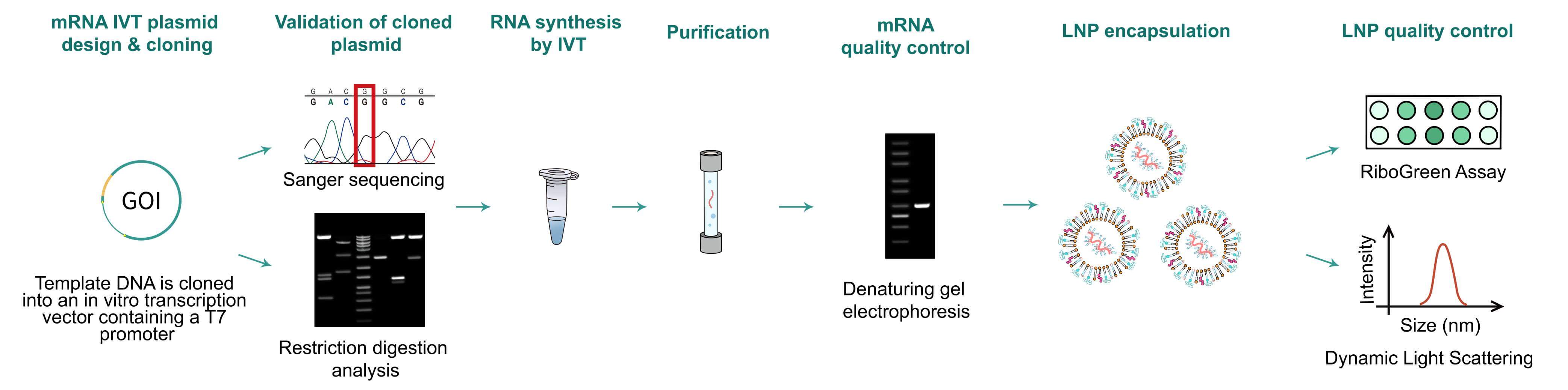
Figure 1. mRNA 합성 및 LNP encapsulation의 일반적인 워크플로우.
- 벡터 클로닝부터 LNP encapsulation까지 최단 5주 소요
- ug에서 gram 스케일까지 최대 10,000nt의 mRNA 및 self-amplifying RNA (saRNA) 합성
- Co-transcriptional 또는 enzymatic capping으로 높은 capping 효율 (최대 99%)
- 면역 반응을 줄이고 in vivo에서 강력한 발현을 달성하기 위한 변형 뉴클레오티드
- m1Ψ
- m5C
- 5moU
- 신속하고 효율적으로 아래와 같은 불순물을 제거하는 독점 정제 기술.
- IVT DNA template
- Partially transcribed mRNA products
- Residual protein
- Double-stranded RNA (dsRNA)
- 일반 및 맞춤형 제형을 사용한 고품질 LNP:
- 높은 encapsulation 효율
- 낮은 polydispersity index (PDI)
- 향상된 안정성
- 증가된 전달 효율
- Antibody-conjugation 적합성
 Quality control (QC)
Quality control (QC)
VectorBuilder는 IVT mRNA 및 LNP encapsulation에 대한 광범위한 QC 방법을 제공합니다. 기본 QC 항목 (√ 표시)은 항상 수행되며, 추가 QC 항목은 개별 프로젝트의 요구사항에 따라 옵션으로 추가될 수 있습니다.
IVT mRNA
LNP encapsulation
| Attribute | QC Assay | Research-grade | GMP-like | |
|---|---|---|---|---|
| Identity | mRNA sequence | Sanger sequencing | √ | √ |
| mRNA length | Denaturing agarose gel electrophoresis | √ | √ | |
| Capillary gel electrophoresis (CGE) | Optional | √ | ||
| General/physical property | mRNA concentration | UV spectrophotometry | √ | √ |
| RiboGreen assay | Optional | √ | ||
| Appearance | Visual inspection | Optional | √ | |
| Potency | Gene expression | In vitro translation followed by Western blot | Optional | Optional |
| Cell transfection | Optional | Optional | ||
| Safety | Sterility | Bioburden test | Optional | √ |
| Mycoplasma | Culture method | Optional | √ | |
| qPCR | Optional | Optional | ||
| Endotoxin | Kinetic chromogenic assay (KCA) | Optional | √ | |
| Purity | mRNA integrity | Denaturing agarose gel electrophoresis | √ | √ |
| Capillary gel electrophoresis (CGE) | Optional | √ | ||
| A260/280 | UV spectrophotometry | √ | √ | |
| Capping efficiency | LC-MS | Optional | √ | |
| PolyA analysis | LC-MS | Optional | √ | |
| Residual dsRNA | Dot blot assay | Optional | √ | |
| Residual plasmid DNA | qPCR | Optional | √ | |
| Residual protein | NanoOrange assay | Optional | √ | |
| Residual solvents | Gas chromatography | Optional | Optional | |
| Attribute | QC Assay | Research-grade | GMP-like |
|---|---|---|---|
| Encapsulation efficiency | RiboGreen assay | √ | √ |
| Particle size, PDI | Dynamic light scattering (Zetasizer) | √ | √ |
| Surface charge | Dynamic light scattering (Zetasizer) | √ | √ |
 기능 검증
기능 검증
- 대용량 클로닝, 합성 및 테스트 플랫폼을 사용하여 GOI 발현에 대한 다양한 서열 최적화 (UTRs, coding sequence, polyA, Kozak 등) 의 효과를 병렬로 평가합니다.
- Antigen presentation, antibody expression, CAR expression 및 CRISPR와 같은 다양한 응용을 위해 확립된 기능 검증 플랫폼.
- 설치류 및 비인간 영장류 (NHP) 를 포함한 동물 모델을 사용하여 LNP-mRNA 유전자 전달 효능 및 안전성을 평가합니다.
기술적인 정보
IVT vector sequence optimization
- UTR
- Coding sequence
- PolyA tail
- Kozak sequence
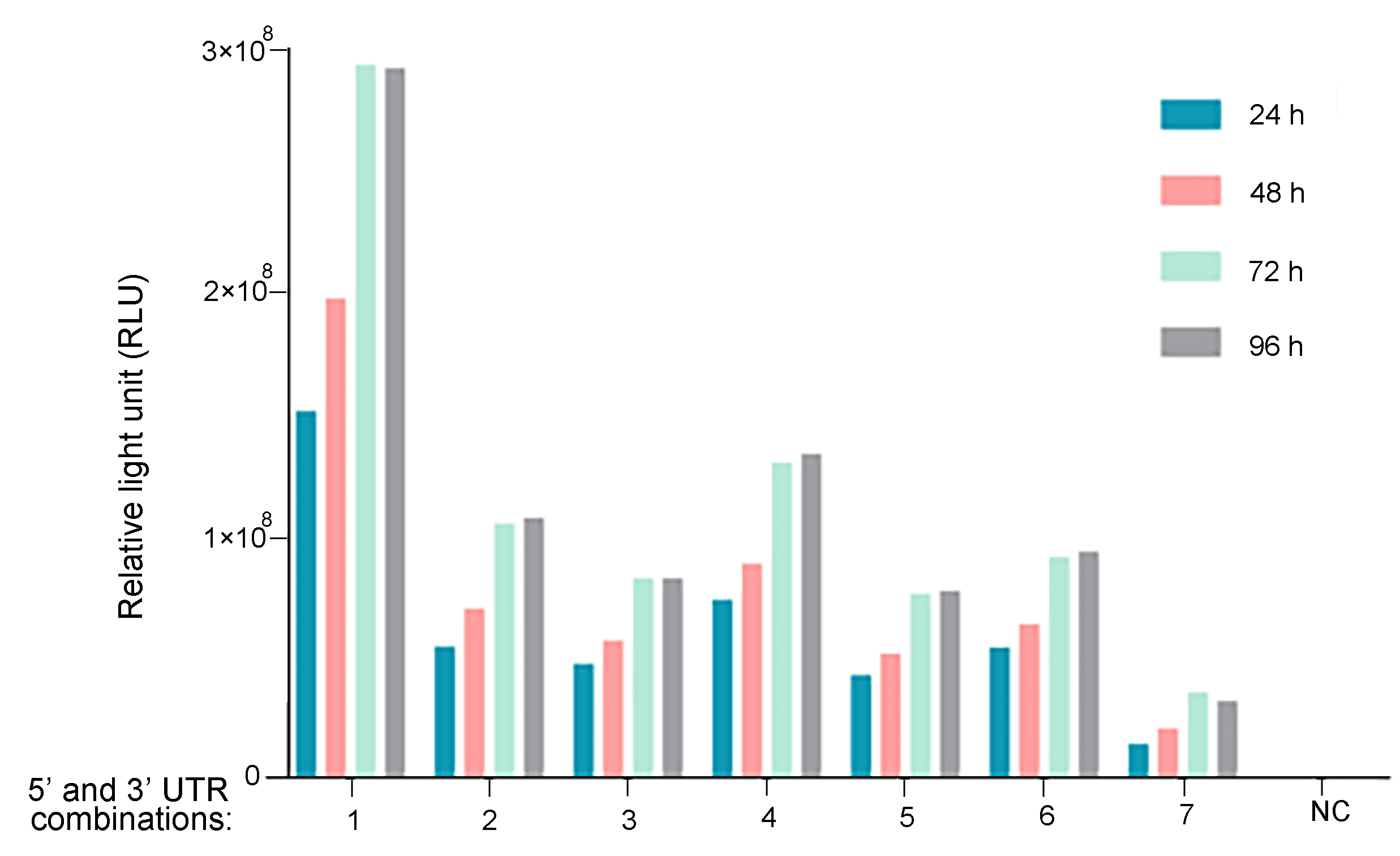
Figure 2. UTR optimization for improved mRNA expression. Different 5' and 3' UTR combinations were tested for regulating Guassia luciferase expression in vitro. 293T cells were seeded on 12-well plates at a density of 2.3x105 cells per well. Cells were transfected with 1 ug of mRNA per well. At 6 h, 24 h, 48 h, 72 h, and 96 h post-transfection, Gaussia luciferase activities were measured from cell culture medium.
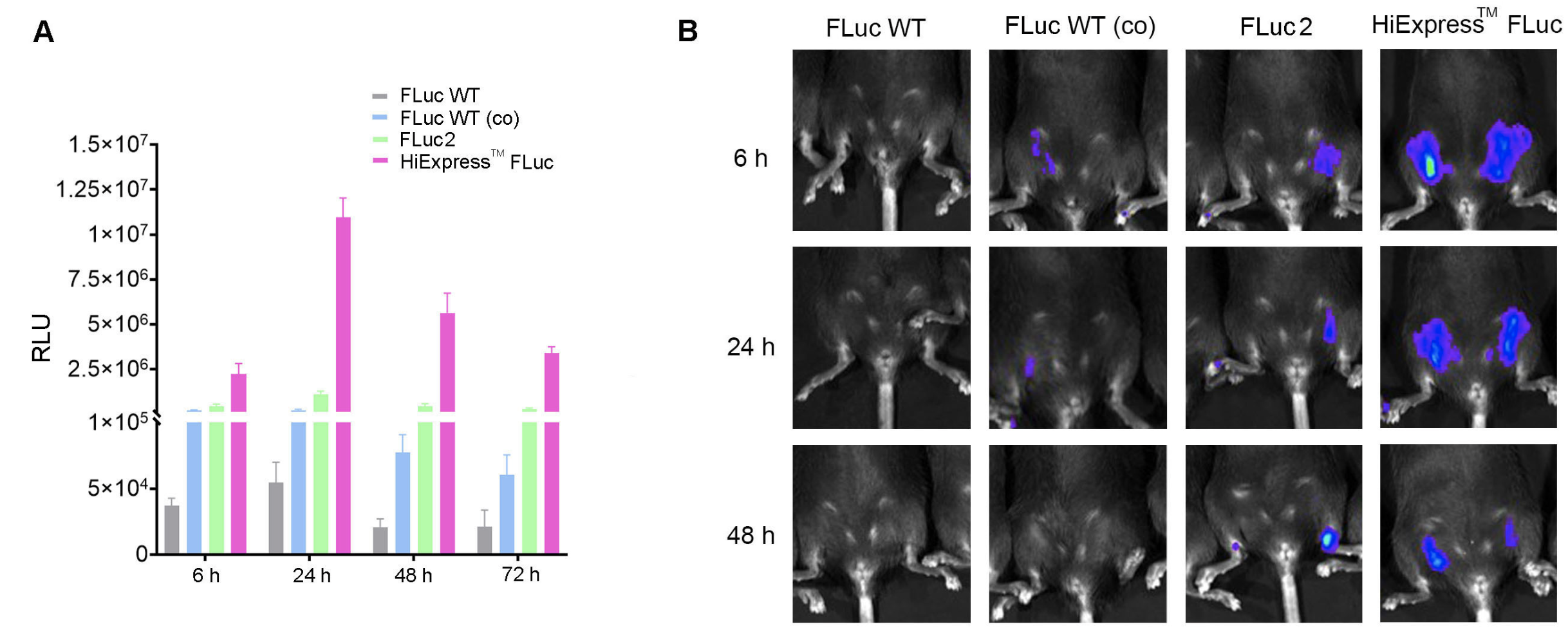
Figure 3. Codon optimization increases mRNA expression in vitro and in vivo. (A) Expression of HiExpress™ Firefly Luciferase mRNA and other luciferase mRNA in HEK293T cells. Cells grown on a 12-well plate were transfected with 0.5 ug of mRNA per well and luciferase activity was measured at 6 h, 24 h, 48 h, and 72 h post-transfection. (B) Luciferase activity measured in adult C57BL/6 mice injected intramuscularly with 30 ug of LNP encapsulated mRNA at 6 h, 24 h, and 48 h post-injection. FLuc WT indicates wild-type firefly luciferase. FLuc WT (co) indicates codon-optimized wild-type firefly luciferase. FLuc2 indicates Luc2 firefly luciferase.
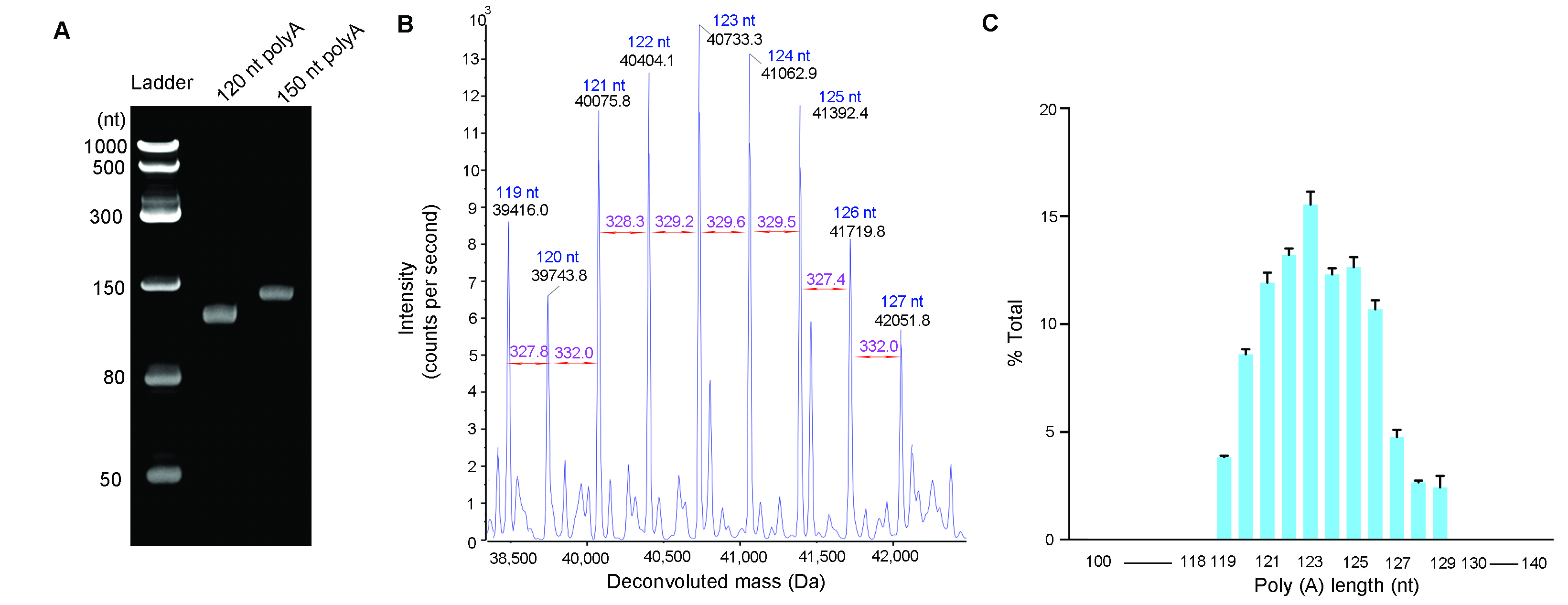
Figure 4. PolyA tail size analysis. PolyA tails were cleaved from IVT mRNA using ribonuclease T1 and isolated by oligo dT affinity chromatography. (A) Isolated polyA tails analyzed by Urea-PAGE gel electrophoresis. 60 ng of polyA tails with expected size of 120 nt and 150 nt were loaded on denaturing Urea-PAGE gel, respectively. (B) Isolated polyA tails analyzed by LC-MS. Deconvoluted spectrum was generated from a 120 pmol of polyA tails with an expected size of 120 nt. (C) Size distribution of the polyA tails with an expected size of 120 nt. The bar graph demonstrates the polyA tails have small heterogeneity. The error bars represent standard deviation from technical triplicates. The weighted average length is 123 nt.
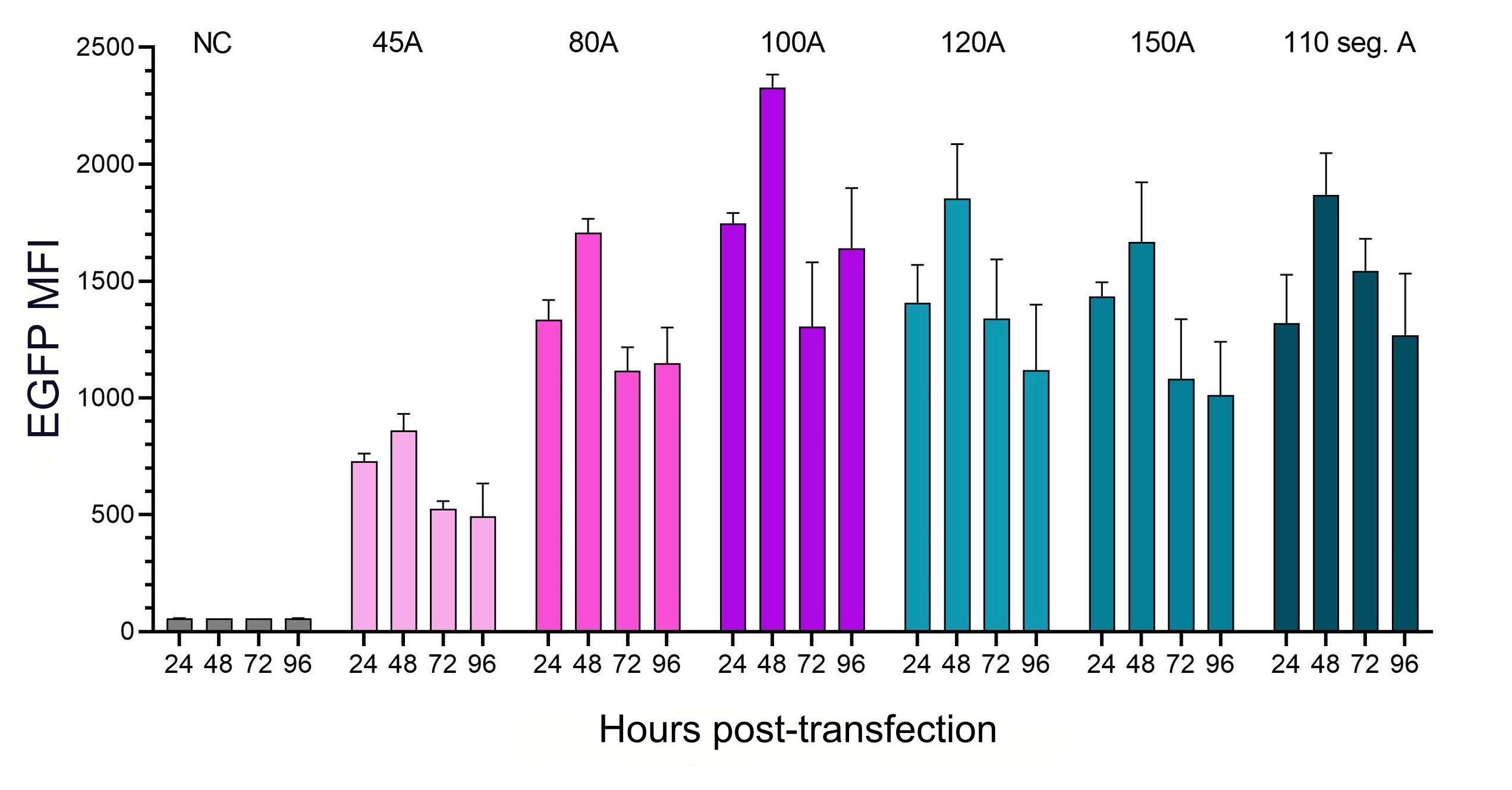
Figure 5. HEK293T cells were transfected with IVT EGFP mRNA with the same Cap1 and UTRs but different polyA tail lengths as listed above.
Figure 6. Kozak sequence optimization increased IVT EGFP mRNA expression in zebrafish. One-cell stage zebrafish embryos were measured for EGFP expression at 6 h post-microinjection of 250 pg of zebrafish EGFP IVT mRNA. Fluorescent images indicated optimized Kozak sequence (version 2) greatly improved EGFP expression in vivo.
IVT mRNA synthesis optimization
- IVT mRNA integrity
- Co-transcriptional and enzymatic capping
- Nucleotide modification
- dsRNA removal
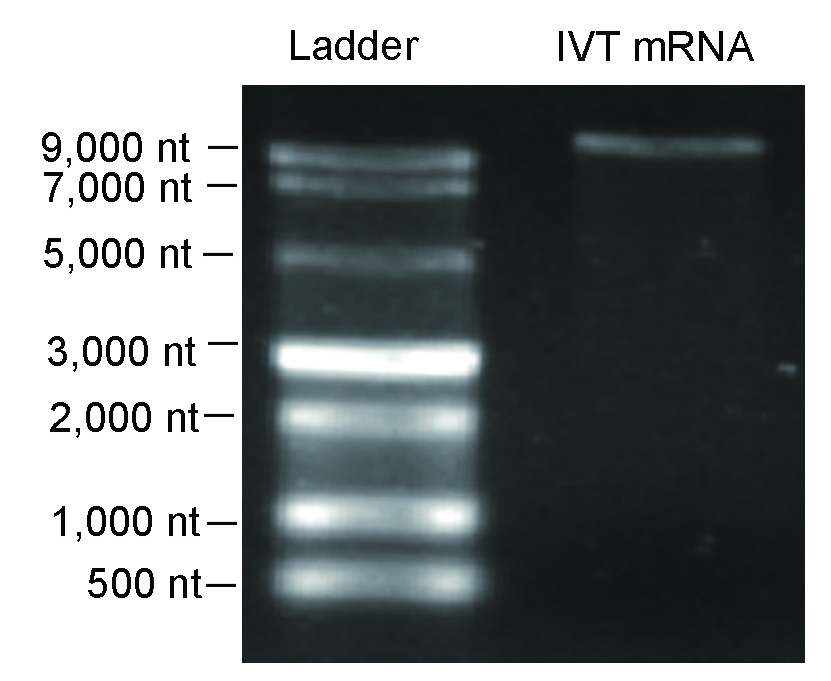
Figure 7. Denaturing agarose gel result indicated high integrity was achieved for >10,000 nt IVT mRNA.
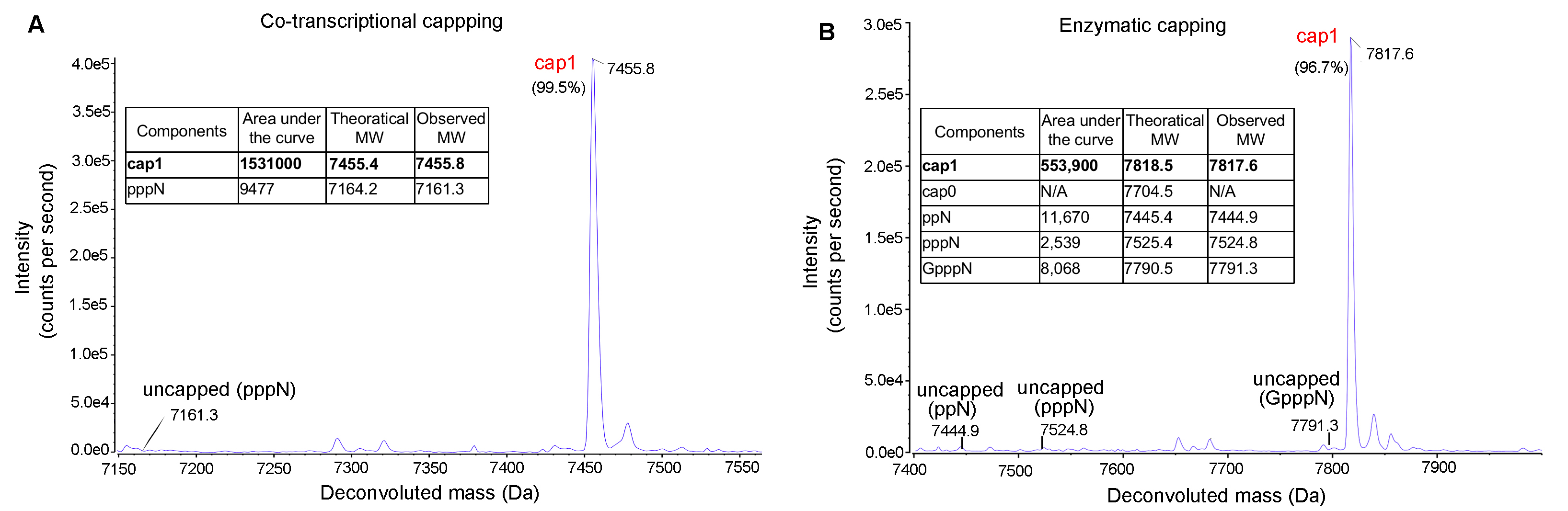
Figure 8. IVT mRNA capping efficiency analyzed by LC-MS. Highly efficient capping (>95%) can be achieved either using (A) co-transcriptional or (B) enzymatic approaches.
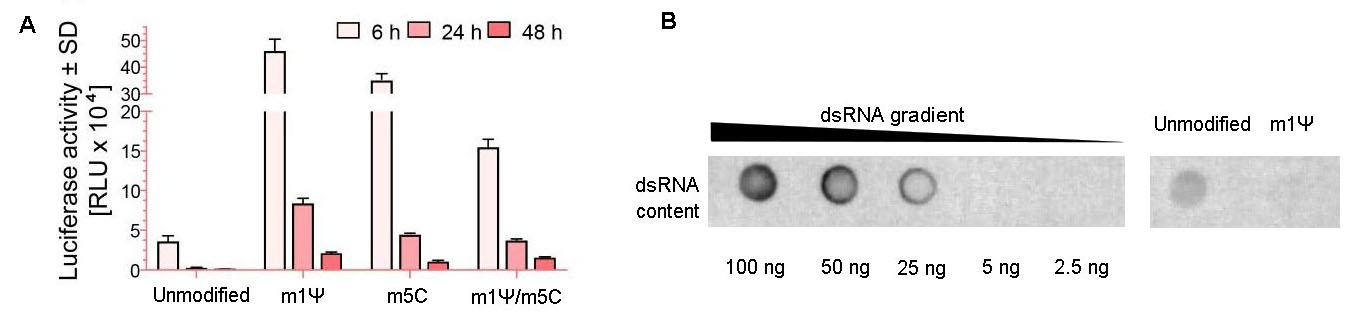
Figure 9. Modified nucleotides increased mRNA expression and decreased dsRNA impurities. (A) Expression of firefly luciferase in 293T cells. The mRNA was generated with or without modified nucleotides, N1-Methylpseudouridine (m1Ψ) and 5-Methylcytosine (m5C). Cells grown on the 12-well plates were transfected with 1 ug of mRNA per well. Luciferase activities in 293T cells at 6 h, 24 h, and 48 h post-transfection were measured. Error bars indicate standard deviations. (B) Equal amount (750 ng per dot) of magnetic bead-purified EGFP IVT mRNA with or without nucleotide modification (m1Ψ) was blotted and subsequently detected by dot blot assay for estimating the dsRNA impurity.
As a major trigger of undesired immunogenicity, double-stranded RNA (dsRNA) is a by-product of IVT. Dot blot result demonstrates extra purification steps (e.g. IP-PR) may be necessary for achieving an ultra-purification scale with very low levels of dsRNA.
Figure 10. dsRNA removal efficiency of different purification processes. Equal amounts (1500 ng per dot) of hSpCas9 IVT mRNA purified by different processes were blotted and subsequently detected by dot blot assay for estimating the dsRNA impurity. Abbreviations: HIC, Hydrophobic interaction chromatography; IP-RP, Ion-pair reversed-phase liquid chromatography.
LNP-mRNA QC data
VectorBuilder has optimized our encapsulation technology to ensure the homogeneity, stability and efficacy of our LNP encapsulated mRNA.
- TEM
- PDI and zeta potential
Cryo-transmission electron microscopy (cryo-TEM) results show good structural integrity and size homogeneity of our LNP-mRNA.
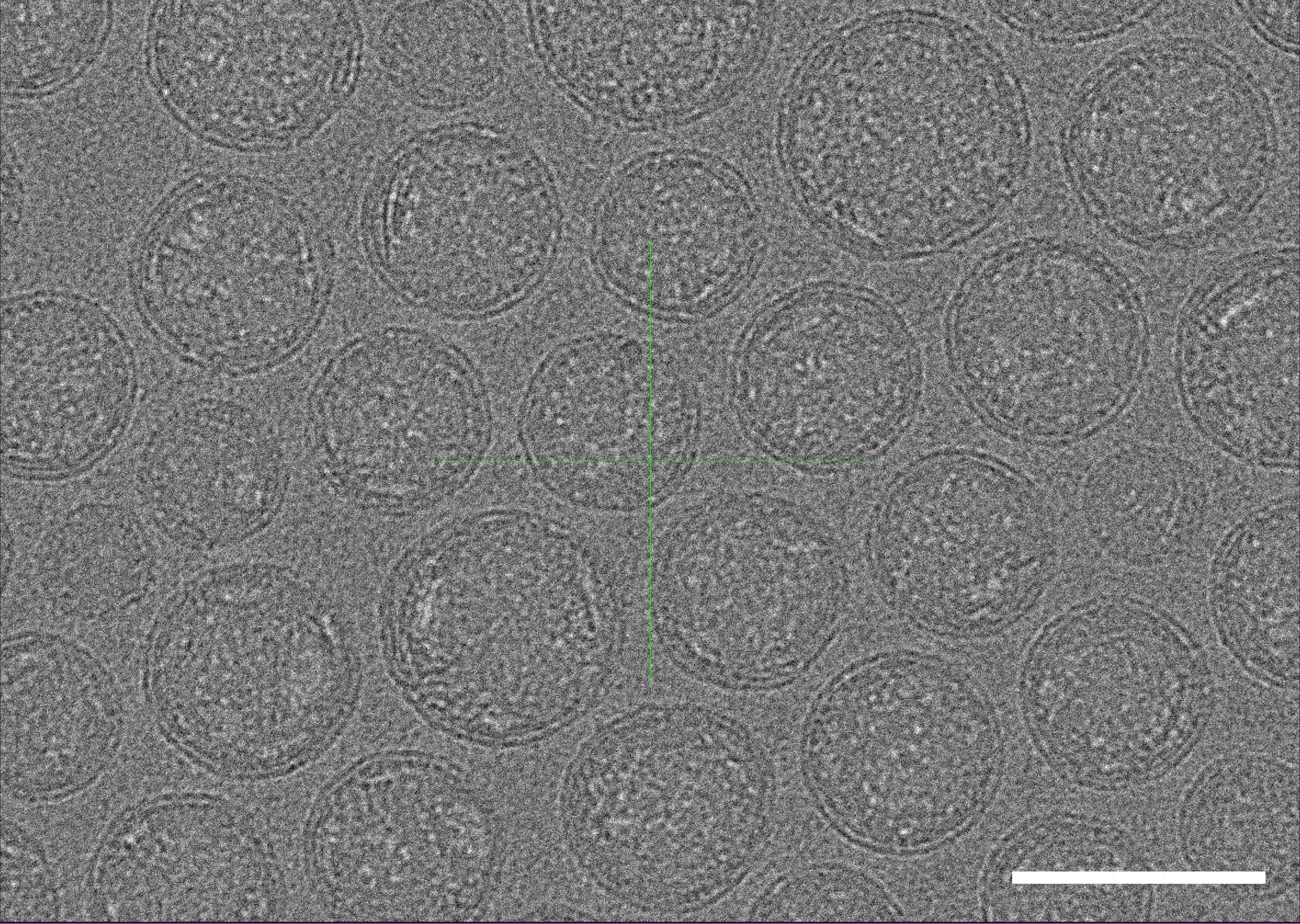
Figure 11. Cryo-TEM micrographs of LNP-mRNA. Scale bar=100 nm.
Dynamic light scattering (DLS) analysis shows our LNP-mRNA products can reach a very low PDI (as low as PDI<0.1).
Figure 12. Particle size and Zeta potential distribution analysis. PDI (A) and Zeta potential (B) was determined by DLS which measures the intensity differences of fluctuated light due to motion of particles. The Zeta potential of the sample is between -1.872 mV and +1.872 mV.
LNP-mRNA functional validation
- LNP-mRNA expression in vitro
- LNP-mRNA expression in vivo
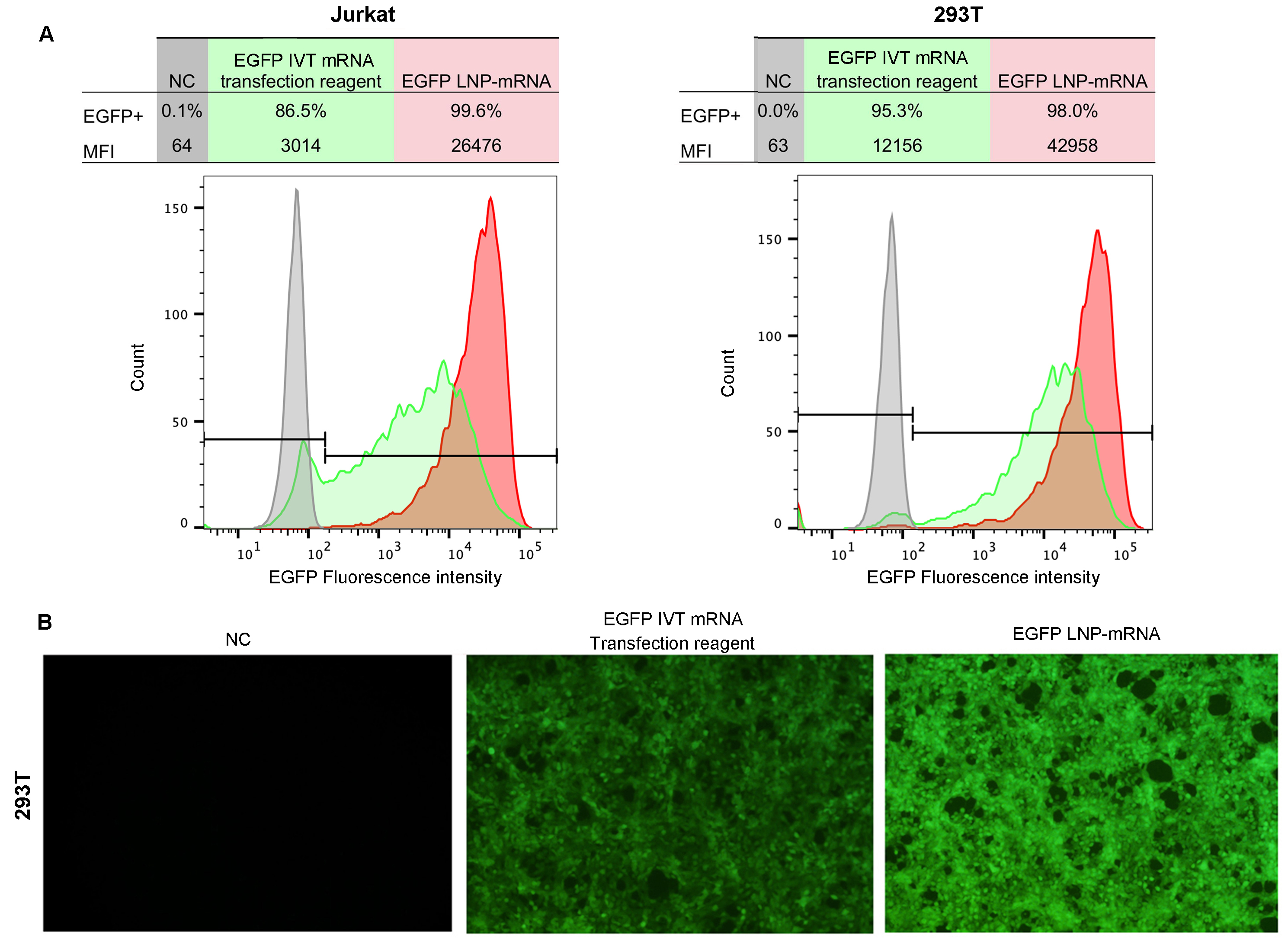
Figure 13. Efficient mRNA delivery and expression using LNP in vitro. Cells were transfected with LNP encapsulated EGFP mRNA or EGFP mRNA mixed with commercial transfection reagent. (A) Flow cytometry analysis of EGFP expression in Jurkat and 293T cells and (B) Fluorescent imaging of 293T cells at 24 hours post-transfection. MFI: median fluorescence intensity.
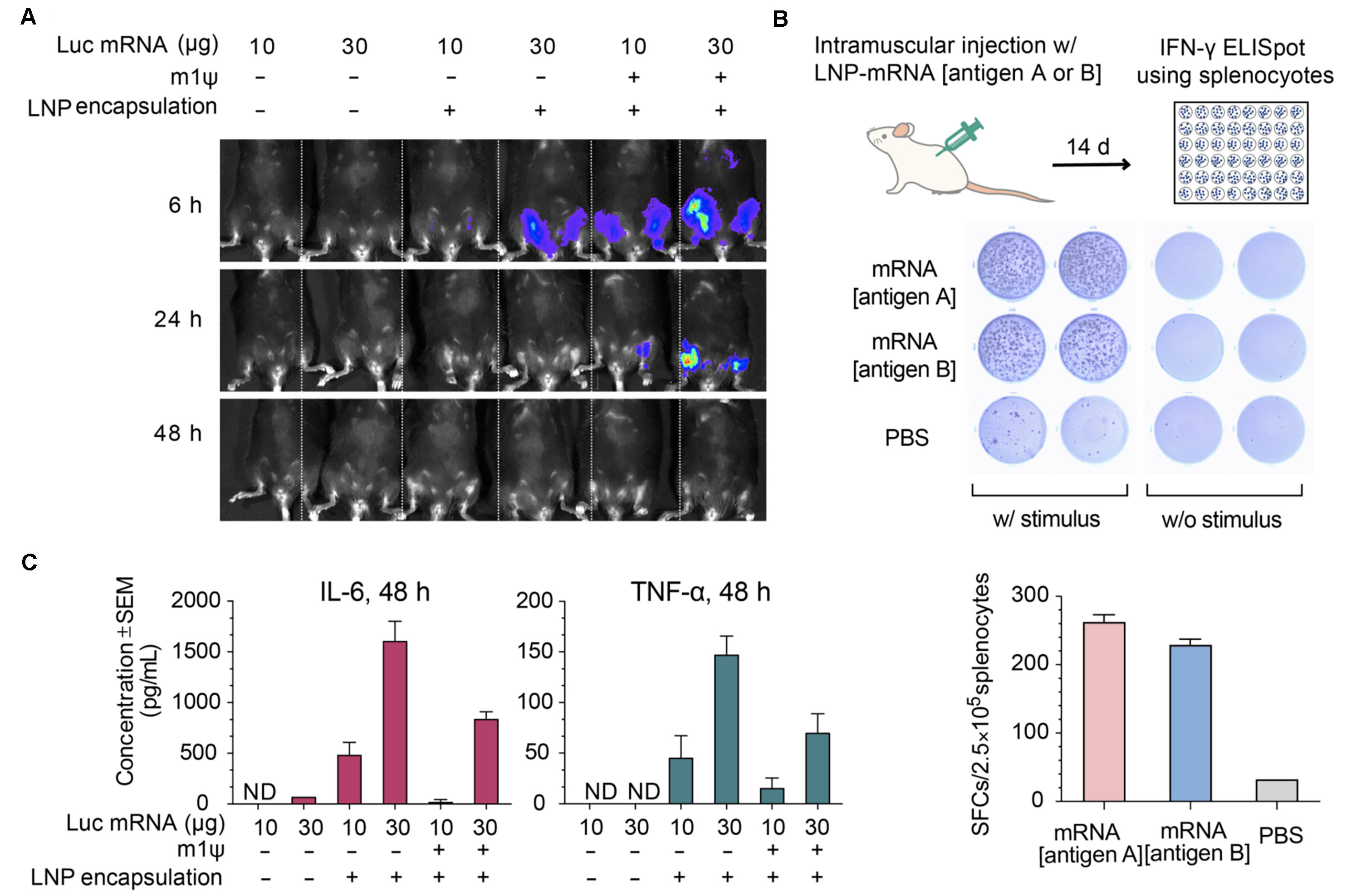
Figure 14. Expression of luciferase (Luc) mRNA and mRNA induced immune response in mice. (A) Luciferase activity visualized by live imaging at 6 h, 24 h, and 48 h post-injection. (B) Two pro-inflammatory cytokines, IL-6 and TNF-⍺, were quantified in the serum at 48 h post-injection. Error bars represent standard errors. Mice strain: C57BL/6J; mice age: 8 weeks; injection method: intramuscular injection. (C) IFN-γ ELISpot assay of splenocytes derived from Balb/C mice 14 days post intramuscular injection of 30 ug LNP-encapsulated mRNA coding for viral antigen A, viral antigen B, or control PBS.
Antibody-conjugated LNP-mRNA
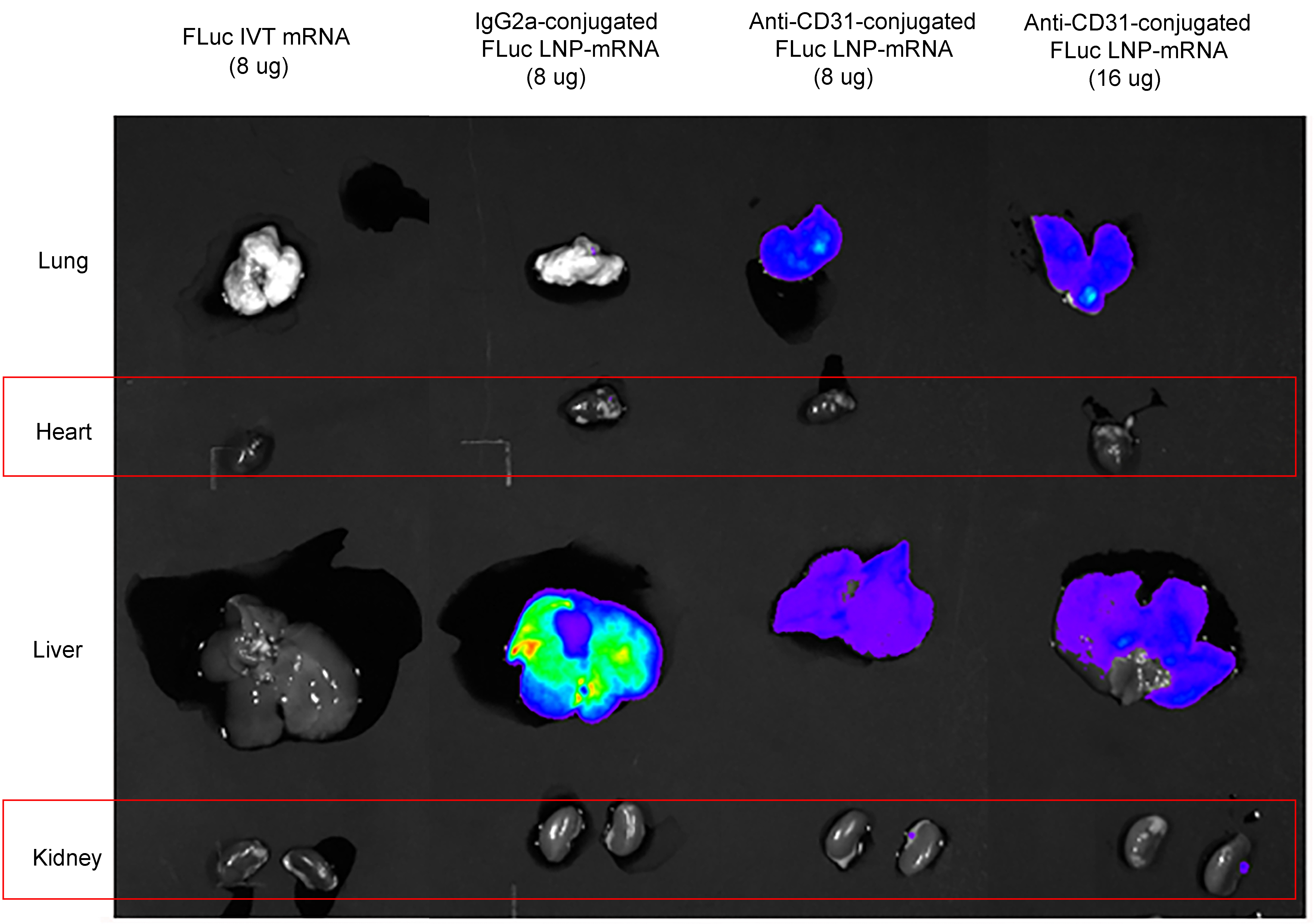
Figure 15. Anti-CD31 conjugated firefly luciferase (FLuc) LNP-mRNA showed improved luciferase expression in lung. Mice strain: C57BC/6J; mice age: 6-8 weeks; mice gender: female; administration route: tail vein. Negative controls: IgG2a-conjugated FLuc LNP-mRNA and naked FLuc mRNA.
문서 자료
물질안전보건자료 (MSDS)시험성적서 (COA)
사용 설명서
- IVT mRNA for in vitro applications
- LNP-mRNA for in vitro applications
- LNP-mRNA for in vivo applications
주문 방법
자주 묻는 질문
Cap 0는 5’ to 5’ triphosphate linkage를 통해 진핵생물 mRNA의 5' 말단에 추가되는 N7-methylguanosine (m7G)을 나타냅니다. 이 변형은 co-transcription으로 발생하는 일련의 효소 반응을 통해 추가되고, nuclear export, 전사체 안정성을 조절하는 기능을 하고, eukaryotic translation initiation factor (eIF4E)에 의한 인식을 통해 mRNA의 translation을 촉진하는 기능을 합니다. Cap 1은 m7G cap 외에 전사된 mRNA 서열의 첫번째 뉴클레오타이드 (m7GpppNm)의 2'O에 메틸기가 추가된 것을 의미합니다. 포유류 세포에서 cap 1 구조는 mRNA가 선천성 면역에 의해 표적화되지 않고 자가로 인식되는 중요한 마커이다. 합성된 mRNA에 cap 1 구조를 추가하면 in vivo에서 mRNA 발현이 향상되고 면역원성이 감소하는 것으로 입증되었습니다.
In vitro transcription된 RNA에 대한 capping은 cap analogs와 함께 co-transcription되거나 또는 효소 반응을 통해 transcription 후에 발생할 수 있습니다. VectorBuilder는 두가지 capping 방법을 모두 제공하며 LC-MS를 사용하여 그 효율성을 검증했습니다. 고객이 선호하는 capping 방법에 따라 IVT mRNA 벡터를 클로닝하기 위한 호환 가능한 백본을 선택합니다.
세포는 외부 RNA를 인식하면 면역 반응을 활성화하는 cytosolic 및 endosomal RNA receptors를 갖고 있습니다. 변형된 뉴클레오타이드는 일반적으로 endogenous cellular RNA에서 발견됩니다. IVT mRNA에 특정 변형된 뉴클레오타이드를 결합하면 면역원성이 감소하고, 2차 구조가 변경되며, 서열 의존적 방식으로 translation 효율과 반감기가 증가합니다. VectorBuilder는 일반적으로 사용되는 N1-Methylpseudouridine(m1Ψ) 및 5-Methylcytosine(m5C)을 포함하여 다양한 변형 뉴클레오타이드를 제공합니다. N1-Methylpsuedouridine 및 5-Methylcytosine은 tRNA에서 처음 확인된 자연 발생 뉴클레오타이드이지만, mRNA를 코딩하는데 사용하는 것을 최근에야 알게 되었습니다. Uridine 과 cytosine의 이러한 메틸화된 유도체는 전통적인 Watson-Crick base pairing을 변경하지 않고 mRNA IVT 및 translation에서 표준 뉴클레오타이드를 대체할 수 있습니다. mRNA 치료제에 사용하는 주요 장점은 RNA immune receptors에 의한 인식을 변경하여 원치 않는 면역 효과를 완화하고 전사체 안정성 및 translation을 향상시키는 능력입니다.









