EtBr 사용: 전기영동된 Gel이 내 샘플을 제대로 반영하는지 어떻게 확신할 수 있습니까?
Gel 전기영동은 DNA의 크기, 구성 및 양을 평가하는 일반적인 방법입니다. 이는 클로닝과 품질관리에서 중요한 단계입니다. 그러나 이론적으로 매우 간단한 이 과정에서 많은 가정과 오해의 소지가 있는 판독값이 나올 수 있습니다.
Gel 전기영동의 기초
분자 발견 초기인 1960년대부터 연구자들은 전류의 영향 하에서 움직이는 전기영동 과정을 이용하여 다르게 이동하는 DNA를 분리해 왔습니다. 그 이후로 분자 생물학 실험실에서 일어난 놀라운 변화에도 불구하고 gel 전기영동은 놀라울 정도로 변하지 않았습니다(Figure 1). 이것은 주로 프로세스의 기술 및 이론적 단순성 때문입니다. 음전하를 띈 DNA는 챔버의 음극에서 양극으로 전류를 따라 용액을 통해 이동합니다(항상 빨간색(+)으로 이동!). 그러나 DNA는 일반적으로 agarose gel 매트릭스를 통해 이동해야 합니다. 작은 DNA 조각은 매트릭스의 구멍을 통해 빠르게 이동하는 반면 큰 DNA 조각은 더 느리게 이동합니다. 100V에서 약 1시간 동안 전기영동 되는 gel에는 일반적으로 짧은 거리를 이동한 큰 DNA 조각과 더 긴 거리를 이동한 작은 DNA조각이 있습니다. 알려진 크기의 band로 구성된 DNA ladder를 사용하여 연구원은 DNA 샘플에 적절한 크기의 DNA가 포함되어 있는지 확인할 수 있습니다. 그런 다음 후속 실험을 위해 DNA band를 잘라내어 분리합니다.
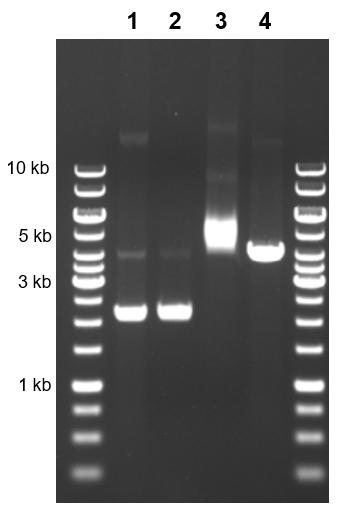
Figure 1. Gel 전기영동 셋업과 결과의 시각화.
교묘한 크기(Size)
Gel 전기영동에서 종종 간과되는 중요한 요소 중 하나는 DNA 조각의 "크기"의 복잡성입니다. 특히 전기영동 중인 샘플이 plasmid DNA인 경우 크기가 DNA 길이(base pairs)와 항상 직접적인 상관관계가 있는 것은 아닙니다. 정확히 동일한 서열을 가진 plasmid도 다양한 형태를 가질 수 있으며, 그 결과 완전히 다른 "크기"가 생겨 gel 상의 위치가 달라집니다(Figure 2). plasmid가 부분적으로 절단되고 한 가닥만 끊어진 nicked plasmid가 생성됩니다. 이 형태에서 plasmid는 완전한 원형을 유지하고 매우 느리게 이동합니다. 제한 효소로 선형화된 plasmid는 gel을 약간 더 빠르게 통과하는 반면, 온전한 원형 DNA는 주어진 시간 내에 훨씬 더 멀리 이동합니다. 그러나 원형 DNA(covalently closed circular DNA 또는 cccDNA라고 함) 내에서도 여러가지 형태가 있습니다. 슈퍼코일 DNA라고 하는 가장 압축된 cccDNA는 양전하 또는 음전하로 감길 수 있으며 양전하 슈퍼코일 DNA는 음전하 수퍼코일DNA보다 약간 작아 gel을 따라 더 이동하게 됩니다.
샘플 내 이런 다양한 형태를 시각화하거나 효소처리를 통해 분리할 수 있습니다. 처리되지 않은 샘플은 모든 형태의 plasmid를 포함할 수 있지만 cccDNA는 T5 exonuclease를 사용하여 분리할 수 있습니다. 그 샘플을 단일 가닥을 절단하고 nicked plasmid를 생성하는 효소인 nickase로 처리할 수 있습니다. 마지막으로 제한효소를 사용하여 선형화된 plasmid를 만들 수 있습니다.
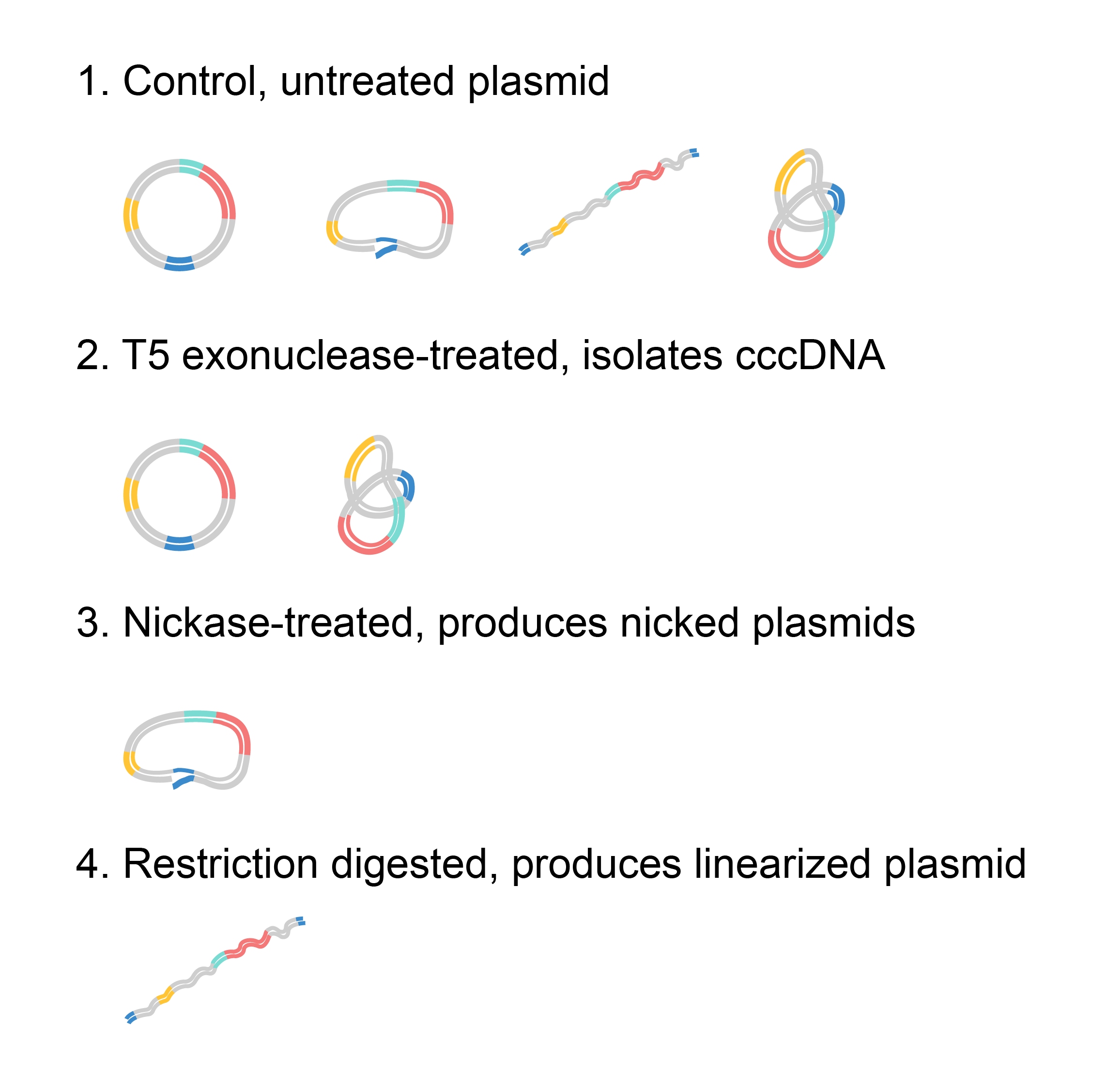
Figure 2. 4180 bp plasmid를 다양한 처리를 한 후 10kb ladder와 함께 전기영동을 함.
Ethidium Bromide에 대한 모든 것
1970년대부터 DNA 샘플과 DNA ladder를 시각화하기 위해 ethidium bromide (EtBr)을 사용하였습니다. EtBr은 DNA에 결합하면 UV 빛 아래에서 강하게 형광을 발하여 gel에서 DNA를 시각화 할 수 있습니다. 그러나 샘플에 결합하는 화합물을 이용할 때, 특히 plasmid DNA와 같은 미묘한 샘플에 대해 고려해야 하는 변수가 추가되는 것입니다. EtBr은 plasmid의 구조에 영향을 미쳐 샘플에 대한 관점을 흐릴 수 있습니다.
EtBr은 일반적으로 0.2-0.5 ug/mL의 농도로 사용되며, 농도가 증가해도 작은 plasmid의 밴드에는 크게 영향을 끼치지 않지만 큰 plasmid는 밴드의 수와 위치에 변형을 줄 수 있습니다. 이것은 EtBr이 DNA 내에 더 많이 껴들어 감으로써 plasmid를 형태로 "고정"하기 때문일 가능성이 높습니다. 예를 들어, EtBr은 슈퍼코일DNA의 형태를 변경하여 음전하 슈퍼코일DNA의 비율을 높이는 것으로 나타났습니다.
EtBr 농도가 샘플 디스플레이에 영향을 미칠 수 있지만 샘플 전기영동 전 DNA와의 상호 작용이 실제 그림을 가릴 수 있습니다. 우리는 전기영동 후 gel을 EtBr로 간단히 처리함으로써 이 문제를 완전히 피할 수 있습니다. EtBr이 첨가된 gel(in-gel)과 전기영동 후 EtBr을 첨가한 gel(post-electrophoresis)로 작은 plasmid를 전기영동하여 비교했을 때 선형화 DNA 및 cccDNA에서 거의 차이가 없었습니다(Figure 3A). 그러나 nicked plasmid는 더 멀리 이동하는 경향이 있는데, 이는 형태를 강화하는 EtBr 삽입 때문일 수 있습니다. 더 큰 plasmid의 경우 in-gel 염색과 전기영동 후 염색 사이에 훨씬 더 중요한 차이를 유발합니다. 대부분의 처리에 대해 밴드의 위치와 수가 모두 변경됩니다(Figure 3B). 중요한 것은 처리 1(대조군)과 처리 2(cccDNA 분리)를 비교할 때 in-gel 염색은 차이가 없는 반면 전기영동 후 EtBr염색은 샘플에서 nicked DNA의 제거를 강조 표시합니다.
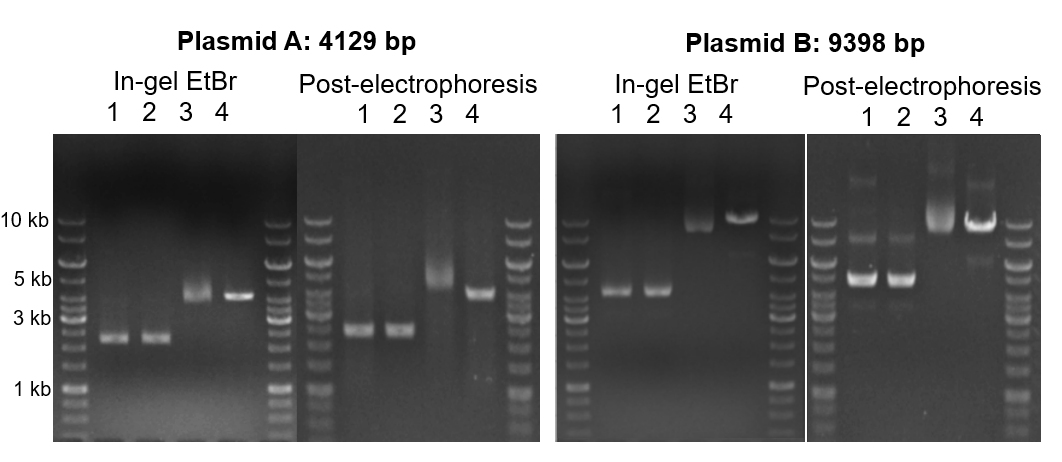
Figure 3. 작은 plasmid(A)와 큰 plasmid(B)의 gel 전기영동은 효소처리 전 과정을 in-gel과 전기영동 후 EtBr 염색방법으로 비교. 1: 처리하지 않은 plasmid ; 2: 효소처리한 plasmid; 3: nickase 처리한 plasmid; 4: 제한효소로 선형화된 plasmid.
고품질 plasmid의 표준은 일반적으로 gel에서 가장 낮은 밴드로 나타나는 슈퍼코일DNA이기 때문에 이러한 불일치를 주목하는 것이 중요합니다. 참고: 원형 단일 가닥 DNA의 희미한 밴드가 때때로 강한 슈퍼코일 밴드 아래에 나타납니다. plasmid를 in-gel 염색 방법으로 분석하면 형태 및 품질에 대한 해석이 편향될 수 있습니다. 특히 T5 exonuclease 처리된 샘플과 비교하여 전기영동 후 EtBr염색으로 본 동일한 plasmid와 효소 처리 결과는 정량화 및 품질 관리를 위해 샘플에 대한 명확하고 편향되지 않은 보기를 보여줍니다. 또한, in-gel 염색과 전기영동 후 염색을 비교하면 cccDNA 내의 형태가 얼마나 유연한지 알 수 있습니다. 이완된 cccDNA는 슈퍼코일DNA로 쉽게 전환할 수 있으며 그 반대도 가능합니다. 따라서 품질을 반영하여 cccDNA가 아닌 슈퍼코일DNA에 초점을 맞추는 것은 EtBr을 포함하여 샘플과 상호 작용하는 일시적인 요인에 너무 높은 중요성을 부여할 수 있습니다.
소규모 클로닝 실험에서 GMP 등급 생산에 이르기까지 샘플을 정확하게 나타내는 gel 결과를 얻는 것이 중요합니다. gel 이 잘못 읽히거나 잘못된 결과가 포함된 경우 후속 실험인 클로닝 및 패키징에 영향을 미칩니다. Gel 전기영동은 수많은 분자 생물학 실험을 위한 기본 도구이므로 성공을 위해서는 기초가 튼튼하고 신뢰할 수 있어야 합니다.
Sources
Aaij C, Borst P. The gel electrophoresis of DNA. Biochim Biophys Acta. 1972 May 10;269(2):192-200. doi: 10.1016/0005-2787(72)90426-1. PMID: 5063906.
Mikhailov VS, Okano K, Rohrmann GF. Specificity of the endonuclease activity of the baculovirus alkaline nuclease for single-stranded DNA. J Biol Chem. 2004 Apr 9;279(15):14734-45. doi: 10.1074/jbc.M311658200. Epub 2004 Jan 21. PMID: 14736888.
Sigmon J, Larcom LL. The effect of ethidium bromide on mobility of DNA fragments in agarose gel electrophoresis. Electrophoresis. 1996 Oct;17(10):1524-7. doi: 10.1002/elps.1150171003. PMID: 8957173.
Stellwagen NC. Electrophoresis of DNA in agarose gels, polyacrylamide gels and in free solution. Electrophoresis. 2009 Jun;30 Suppl 1(Suppl 1):S188-95. doi: 10.1002/elps.200900052. PMID: 19517510; PMCID: PMC2757927.
Stettler UH, Weber H, Koller T, Weissmann C. Preparation and characterization of form V DNA, the duplex DNA resulting from association of complementary, circular single-stranded DNA. J Mol Biol. 1979 Jun 15;131(1):21-40. doi: 10.1016/0022-2836(79)90299-7. PMID: 490644.



